Resumen
La comunicación intercelular es esencial para el desarrollo de células, tejidos y órganos especializados y es fundamental en una variedad de enfermedades, incluido el cáncer. El conocimiento actual establece que los diferentes tipos de células se comunican mediante interacciones ligando-receptor: las hormonas, los factores de crecimiento y las citoquinas. Se liberan en el espacio extracelular y actúan sobre los receptores, que a menudo se expresan de una manera específica según el tipo de célula. Los RNAs no codificantes (ncRNAs) están surgiendo como factores de comunicación en estados fisiológicos y patológicos. Estas clases de ARNs abarcan microARN (miARN, reguladores postranscripcionales de la expresión génica bien estudiados), ARN largos no codificantes (lncRNAs) y otros ncRNAs. Los ARN largos no codificantes son diversos en longitud, secuencia y estructura (lineal o circular), y sus funciones se describen como regulación transcripcional, inducción de cambios epigenéticos e incluso regulación directa de la actividad de la proteína. También se ha informado que actúan como esponjas de miARN, que interactúan con miARN y modulan la disponibilidad a mARN. Es importante destacar que los lncRNAs pueden tener un patrón de expresión específico de tipo celular. En este artículo, proponemos que las interacciones lncRNA-miRNA, análogas a las interacciones receptor-ligando, son responsables de los resultados específicos de tipo celular. La unión específica de los miRNAs a los lncRNAs puede conducir a las cascadas de señalización específicas del tipo celular y modular los circuitos de retroalimentación bioquímica que en última instancia determinan la identidad celular y la respuesta a los factores de estrés.
Abstract
Intercellular communication is indispensable for the development of specialized cells, tissues and organs and its deregulation is implicated in a variety of diseases, including cancer. Current knowledge states that different cell types communicate through ligand-receptor interactions: hormones, growth factors and cytokines. They are released into the extracellular space and act on the receptors, which are often expressed in a specific way according to the type of cell. The non-coding RNAs (ncRNAs) are emerging as communication factors in physiological and pathological states. These classes of RNAs encompass microRNA (miRNA, well studied post-transcriptional regulators of gene expression), long non-coding RNAs (lncRNAs) and other ncRNAs. The long non-coding RNAs are diverse in length, sequence and structure (linear or circular), and their functions are described in transcriptional regulation, induction of epigenetic changes and even direct regulation of protein activity. They have also been reported to act as miRNA sponges, by interacting with miRNAs and modulating their availability towards mRNAs. It is important to note that the lncRNAs may have a tissue and cell-type specific expression pattern. In this article, we propose that lncRNA-miRNA interactions, analogous to receptor-ligand interactions, are responsible for cell-type specific outcomes. The binding of the miRNAs to lncRNAs can lead to cell-type specific alterations in signaling cascades and modulate the biochemical feedback loops that ultimately determine cell identity and response to stress factors.
Palabras clave: Cáncer; Comunicación celular; Vía de señalización celular; ARN largo no codificante (ARNnc); MicroRNA (miR); Epigenética.
Keywords: Cancer; Cellular communication; Cell signaling; Long non-coding RNA (IncRNA); MicroRNA (miR); Epigenetics.
INTRODUCCIÓN
Uno de los primeros científicos que habló del núcleo de las células y de la sustancia que había en su interior, que llamó nucleína fue Miescher en el año 1869. En décadas posteriores se avanzó muy poco en el conocimiento de dicha sustancia hasta el descubrimiento del DNA en 1950, en el que se evidenció que el DNA llevaba todas las instrucciones de las células y que se transmitían de las células parentales a las células hijas. Posteriormente en el año 1953 Watson y Crick descubrieron la estructura del DNA utilizando las notas del laboratorio y gráficos de la Dra. Rosalind Franklin. En los siguientes años destacan los avances de Sanger, en los que describió el primer método para secuenciar las moléculas de DNA y que fue un gran punto de inflexión para el conocimiento del DNA (1). 2 años más tarde científicos de Genentec pudieron modificar genéticamente un organismo y producir somatostatina, insulina y hormona de crecimiento humana en microorganismos modificados. Cuatro años después, 1983, Kary Mullis publicó el primer artículo describiendo la técnica de la polimerasa, o PCR. En 1984 comenzó el proyecto del genoma humano, que culminó en el año 2003. Es de destacar asimismo que en 1990, la FDA aprobara el primer experimento de terapia génica en los EEUU y que el primer clonaje de un mamífero, la oveja Dolly, fue en el año 1996.
EL PARADIGMA DE LA COOPERACIÓN CLONAL EN LA CARCINOGÉNESIS
Inicialmente se pensaba que la transformación tumoral era a partir de una única célula que iba acumulando mutaciones genéticas. Al final aquellas células más agresivas son las que iban a ser invasivas y capaces de dar metástasis (2). Hoy en día muchos grupos consideran que en la formación de un tumor maligno es necesaria la cooperación clonal de más de una célula maligna o clones tumorales malignos (3-5). Es difícilmente imaginable que una única célula pueda acumular las alteraciones genéticas que se requieren para que tengan todas las propiedades de agresividad y malignidad, incluyendo resistencia a apoptosis, inducir factores angiogénicos, evitar la senescencia, generar sus propias señales de crecimiento….. Por tanto, en dicha cooperación clonal entre células, se tienen que complementar dichas vías descritas por Hanahan y Weinberg (6), hace ya dos décadas. Por tanto, la cooperación clonal conlleva que las células tienen que comunicarse entre sí y liberar sustancias y factores para que el conjunto de células pueda exhibir los rasgos de malignidad. La comunicación celular es a través de factores secretados directa o indirectamente, vía exosomas u otras vesículas extracelulares. Este concepto de comunicación y cooperación clonal ha sido demostrado en la formación de las metástasis. En diversos trabajos se ha observado cómo en las metástasis se observan varios tipos de clones y que sólo aquellos clusters celulares complementarios son capaces de formar metástasis (7). Por tanto, cortar la comunicación celular entre dichos clones abre unas perspectivas clínicas francamente interesantes a corto y medio plazo.
PARADIGMAS ACTUALES DE LA GENÉTICA MOLECULAR
Hoy en día nuestro conocimiento se basa en el estudio del DNA codificante, es decir aquellos genes que dan lugar a un RNA mensajero, el cual se traduce en una proteína que es la que ejerce la función final. Estas dos acciones fisiológicas se denominan transcripción de DNA a RNA y traducción de RNA a proteína. Por tanto, muchas de las enfermedades actuales se basan en alteraciones genéticas a nivel del DNA o de la síntesis de las proteínas asociadas junto con alteraciones que pueden ser como mutaciones en el DNA, translocaciones, amplificaciones génicas, pérdidas de secuencias génicas, etc. Asimismo son de gran relevancia las alteraciones epigenéticas en las que hay alteraciones de la transcripción de DNA a RNA generalmente por silenciamiento a nivel del promotor por metilación del mismo o por alteración de las histonas o proteínas que regulan la envoltura del DNA (1).
Pero este paradigma cambió levemente en el año 1993, con la concesión del Premio Nobel a Sharp y Roberts, que describieron que la transcripción de DNA a RNA no es tan sencilla o directa, sino que ellos demostraron que hay variantes en dicha transcripción, con formación de múltiplos RNA’s a partir del mismo DNA. Es lo que se llama “splicing”(8). Dicha alteración de la transcripción conlleva gran variabilidad de proteínas. Por ejemplo, variantes del receptor de andrógenos con proteínas anómalas o truncadas, pueden encontrarse en carcinomas de próstata en los que todavía mantienen actividad y no pueden ser contrarrestadas con los fármacos actuales.
LOS NUEVOS CONCEPTOS DEL RNA NO CODIFICANTE
En la actualidad se han descrito en torno a 20.000 genes que codifican para proteínas, que corresponden sólo entorno al 2% del genoma. El resto del DNA son tránscitos que hasta hace pocos años se han considerado como DNA basura, ruido, o restos que se heredaban sin ninguna función conocida o determinante (9).
Dicho DNA no codificante o “basura” constituye una parte muy pequeña del genoma de las bacterias y su proporción va aumentando hasta los humanos, siendo en torno al 5% en bacterias, 15-20% en levaduras, más del 50% en gusanos y casi el 98% en humanos. De dichos RNA’s no codificantes se están describiendo diversos tipos, siendo los más representativos los microRNA’s, de los cuales se han descrito casi 2.000 en secuencias diferentes, de los antisens RNA más de 3.200, de los denominados small nucleolar RNA’s más de 1.500 y los denominados long coding RNA’s, de los que en la actualidad se conocen más de 5.000 (10).
Los microRNA’s son secuencias de ARN monocatenarios de entre 9 y 25 nucleótidos de longitud. Están implicados en la regulación génica post-transcripcional y probablemente también en la metilación del DNA. Actúan fundamentalmente unidos en las regiones 3’ de los RNA mensajeros de genes codificantes, y al unirse, lo que hacen es impedir su traducción en proteínas y por tanto, inhibir su función. Por tanto, la presencia de microRNA’s en la mayoría de las situaciones conlleva el que los genes codificantes, a través de sus mRNA’s canónicos, no puedan producir las proteínas correspondientes.
La alteración de los microRNA’s se está describiendo en muchos procesos de enfermedades humanas, tanto a nivel de metabolismo, sistema inmunológico, patología tumoral, e incluso a nivel fisiológico durante el desarrollo. Existe una gran variabilidad entre los diversos tipos celulares y los tumores, habiéndose publicado miles de artículos describiendo alteraciones de los mismos en muchos tipos de enfermedades.
En resumen, los microRNA’s pueden controlar lo que es la formación de las proteínas con un doble mecanismo. Por un lado, mecanismo supresor, es decir, inhibiendo la formación de la proteína. Pero también pueden tener un efecto protumoral u oncogénico cuando inhiben a un microRNA que estaba evitando síntesis de un gen supresor tumoral.
El paradigma por tanto de las vías de señalización canónicas, en las que hay varias proteínas que van transmitiendo una señal de una a otra, se modifica enormemente con la presencia de microRNA’s que pueden formar cortocircuitos en la transmisión de la señal de una proteína a otra, con los denominados feed-backs positivos o negativos. Por tanto, la regulación de la traducción de señales es mucho más compleja de lo que se pensaba inicialmente.
Los denominados long non coding RNA son secuencias de más de 200 pares de bases de longitud que pueden en ocasiones codificar proteínas de muy bajo peso molecular. Estos RNA’s pueden tener también un efecto regulador epigenético a través del control de la remodelación de la cromatina y también a nivel del splicing de mRNA’s mensajeros, de factores de transcripción y especialmente a nivel de la estabilidad de los mRNA’s mensajeros. Por tanto esta familia de más de 5.000 miembros de ácidos nucleicos tienen múltiples funciones, muchas de ellas desconocidas en la actualidad. Son importantes a nivel de las secuencias teloméricas, a nivel de la inactivación del cromosoma , y de una forma muy interesante, el control de muchos microRNA’s. Los long non coding RNA son capaces de atraer como “esponjas” algunos microRNA’s e inhibir su función (Fig. 1).
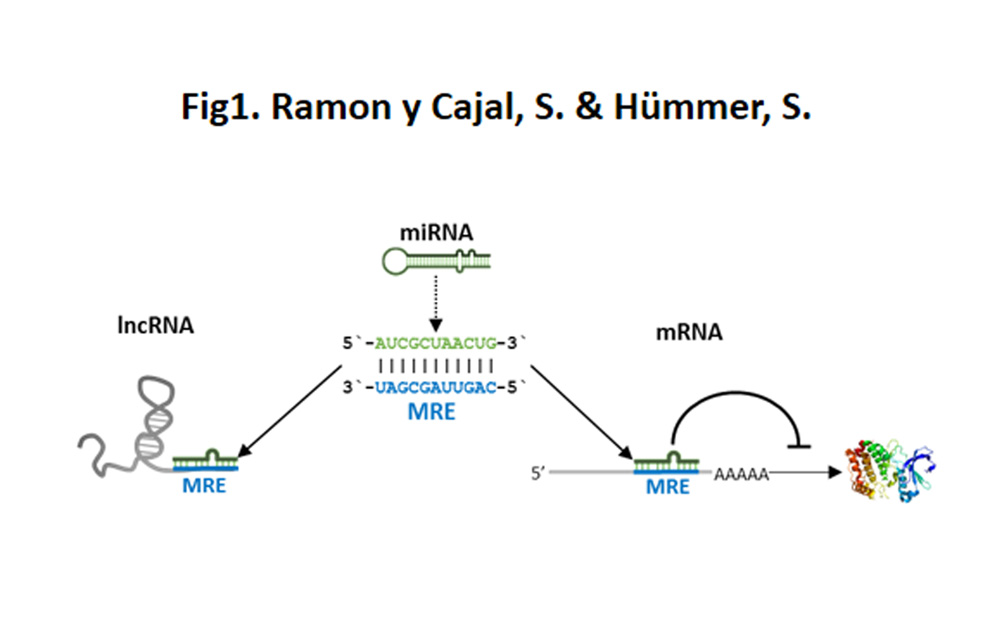
La descripción y conocimiento funcional de estos long non coding RNA, está complicando el conocimiento de las enfermedades donde hay muchos factores que puedan regular la transmisión de las señales en un sentido o en otro, siendo ineludible por tanto la introducción de técnicas o aproximaciones como las de biología der sistemas u otros abordajes bioinformáticos.
LONG NON CODING NG RNA Y ESPECIFICIDAD CELULAR ARN LARGOS NO CODIFICANTES
Todos los ARN de más de 200 nucleótidos que no se traducen en proteínas se clasifican colectivamente como ARN largos no codificantes (ARNnc) (11-13). De manera similar a los ARNm, los ARNnn son transcritos por la ARN polimerasa II y con frecuencia están sujetos a modificaciones post-transcripcionales, como el límite de 5 ‘, la poliadenilación y el empalme de 3’ (14). Aunque menos estudiados que los miRNA, ya se han descrito varios miles de lncRNAs y, gracias a los avances técnicos en las técnicas de secuenciación de RNA y los métodos de predicción computacional, el número total de lncRNAs identificados sigue aumentando (14,15). Definidos únicamente por su longitud, los lncRNAs constituyen la clase más grande de ncRNAs en el genoma de los mamíferos, y pueden categorizarse aún más en ncRNAs intergénicos largos (lincRNAs), RNAs antisentido (asRNAs), pseudogenes y RNAs circulares (circRNAs) (14,16). Los ARNnc cumplen una variedad de funciones al interactuar con el ADN, el ARN y las proteínas, y pueden describirse según su mecanismo de acción hacia su molécula interactiva como potenciadores, señuelos, guías o andamios (17).
El primer análisis de agrupamiento no supervisado de transcripciones individuales en diferentes tejidos reveló que el 78% de los ARNnc (en comparación con el 19% de los ARNm) se expresaron de una manera específica del tejido (18). A medida que avanzaban las técnicas de secuenciación, esta especificidad de expresión se había observado a nivel de células individuales, e incluso se había observado una expresión diferencial de célula a célula por (19); Podríamos confirmarlo en nuestros propios estudios (no publicados) en la línea celular de cáncer de mama triple negativo MB-MDA231. En el campo de la biología del cáncer, el perfil de expresión de lncRNA se ha propuesto recientemente como un fuerte factor pronóstico e incluso como un objetivo terapéutico (20).
A pesar del creciente catálogo de lncRNAs, la mayoría de los productos de transcripción detectados permanecen funcionalmente sin anotar. La predicción de la función génica basada en la homología de secuencia para genes codificantes de proteínas es un reto (21). Por lo tanto, la clasificación de ARNnc requiere herramientas computacionales especializadas o estudios funcionales de todo el genoma como se realiza actualmente mediante el uso de técnicas de eliminación de genes (por ejemplo, CRISPERi) (22).
lncRNAs EN CÁNCER
La expresión diferencial de lncRNAs se ha descrito en una variedad de afecciones patológicas que incluyen enfermedades cardiovasculares, autoinmunes, neurodegenerativas y particularmente en cáncer (23-26). El primer lncRNA reportado con una expresión aberrante en el cáncer fue el cáncer de próstata asociado a 3 (PCA3) (27), que se identificó mediante el análisis de visualización diferencial de las transcripciones en el cáncer de próstata humano normal. PCA3 fue el primer biomarcador basado en lncRNA aprobado por la FDA para su uso en la práctica clínica. Desde entonces, ha demostrado ser una prueba útil y no invasiva para el cáncer de próstata (28). En años posteriores, muchos otros lncRNAs han sido identificados por tener un valor altamente predictivo en el diagnóstico de diferentes tipos de cáncer. Entre ellos, la expresión desregulada del lncRNA HOTAIR, originalmente descrita en el cáncer de mama, está asociada con la progresión del cáncer en 26 tipos de tumores humanos (29,30). Recientemente, la aplicación de la secuenciación de próxima generación en una variedad de transcriptomas de cáncer diferentes descubrió miles de lncRNAs con expresión aberrante en diferentes tipos de cáncer (31). Estas cifras aumentan aún más cuando se toman en cuenta los polimorfismos de un solo nucleótido (SNP) que están asociados con el cáncer (32).
Con respecto a la metástasis, algunos lncRNAs se han asociado con tumores metastásicos más agresivos e incluso con colonización de células cancerosas en sitios de órganos específicos (33). Por ejemplo, en el carcinoma colorrectal, la expresión del ARNnc CCAT2 se correlacionó con una mayor incidencia de metástasis hepáticas (34). Otro ejemplo es la asociación de niveles elevados de HOTAIR con una mayor incidencia de metástasis hepáticas en el cáncer gástrico (35) y con metástasis cerebrales en el carcinoma pulmonar de células no pequeñas (36).
A pesar de la enorme cantidad de lncRNAs descritos como que tienen una expresión aberrante en diferentes tipos de cáncer, hasta la fecha, solo unos pocos se han caracterizado funcionalmente [revisado en (31)]; los estudios existentes han identificado las funciones supresoras de tumores y oncogénicas de lncRNAs (31,37).
INTERACCIÓN lncRNA Y miRNA
La evidencia reciente indica que los lncRNAs pueden regular la expresión de genes en trans actuando como “esponjas” de miRNA (38-41). Estos lncRNAs reguladores de miRNA pertenecen a un grupo de RNA codificantes y no codificantes que se denominan colectivamente ceRNA (competidor de RNA endógeno) (40,42). La unión de miRNAs a lncRNAs se logra a través de MREs, los mismos elementos de secuencia que se usan para las interacciones de miRNA y mRNA. Los ceRNAs contienen MRE para uno o varios miRNAs, y se cree que la unión aísla a los miRNAs y, por lo tanto, permite la traducción de dianas miRNA endógenas (Fig. 1).
En los últimos años, este concepto se ha descrito para la expresión de diferentes genes implicados en la progresión tumoral. Un ejemplo de un lncRNA oncogénico es UCA1, que controla la disponibilidad de miR-18A y, por lo tanto, determina la expresión del oncogen YAP1 (43) (Fig. 2A). En contraste, el gen supresor de tumores PTEN está regulado por el ARNnc CCAT2 al actuar como un ARN competitivo para miR-21 (Fig. 2B) (44). En los últimos años, se han descrito muchos más ceRNAs en el cáncer.

Además de secuestrar los miRNAs, también se ha informado que los lncRNAs compiten con los miRNAs al unirse directamente a los mRNAs. Se ha informado que los miRNAs inducen desestabilización de lncRNAs, sin embargo, algunos lncRNAs contienen precursores de miRNAs (45). Esto sugiere una interacción compleja entre lncRNAs y miRNAs, que en última instancia determina la estabilidad y la traducción de mRNAs que codifican proteínas. En particular, estudios recientes han revelado que los ceRNAs tienen funciones importantes en la patogénesis del cáncer (46,47). Por ejemplo, las alteraciones en la expresión de factores clave en las vías de señalización oncogénicas, como BRAF, se han relacionado con cambios en el nivel de ceRNAs (48,49). Estamos empezando a comprender estas complejas interacciones moleculares, su lugar en las redes reguladoras funcionales que controlan los procesos celulares y sus implicaciones en el cáncer (50).
SITUACIÓN DEL PROBLEMA ACTUAL
En primer lugar, estamos ante un gigantesco puzle y caos molecular, según los paradigmas actuales. Hay cientos o miles de alteraciones moleculares en los tumores malignos. Como se mencionaba, más de 2.000 alteraciones genéticas constitutivas ya se han descrito, a nivel de genes codificantes, incluyendo miles de translocaciones. Ahí hay que sumar el papel funcional y patogénico de los microRNA’s y de los long non coding, así como de las alteraciones epigenéticas. Todo ello hace que estemos ante un auténtica sopa de letras en la mayoría de los casos, cuando se realizan estudios completos de expresión génica, de proteómica, de metabolómica o de factores de señalización celular.
Además, el paradigma molecular está promoviendo numerosos ensayos clínicos, con la idea que inhibición de determinadas alteraciones oncogénicas pueden tener un efecto beneficioso para los pacientes. Son ensayos clínicos denominados tipo Basquet, en los que se trata a los pacientes en función de las alteraciones genéticas que puedan ser inhibidas. No obstante, hay que reseñar que no siempre conllevan una mejor respuesta al tratamiento. Por ejemplo, las mutaciones del oncogén B-RAF características de más de un 50% de los melanomas, responden positivamente a inhibidores específicos durante meses o incluso algunos años. Por el contrario, en los adenocarcinomas de colon con la misma mutación y tratados con los mismos inhibidores, la respuesta es negativa, observándose generalmente una reactivación del tumor.
PROPUESTA QUE PLANTEAMOS
En las vías de señalización oncogénicas clásicas (PI3K / AKT / mTOR o RAS / MAPK), deben existir capas adicionales de regulación, para explicar los procesos específicos de tejidos y órganos. Nuestra hipótesis es que la específicidad según tipo celular puede ser mediada por la interacción entre lncRNAs y miRNAs. Sobre la base del concepto de ceRNAs, la expresión específica según tejido y células de los lncRNAs podría ser un mecanismo clave para apoyar la regulación específica de las vías de señalización. En este sentido, el perfil de traducción (es decir, el proteoma) de los ARNm regulados por miARN puede regularse mediante la expresión específica de tipo celular de los “long non coding RNA”. Por tanto, la interacción entre lncRNAs y miRNAs podría afectar las cascadas de señalización al modular la activación de proteínas dentro de las vías de una manera específica (Fig.3).
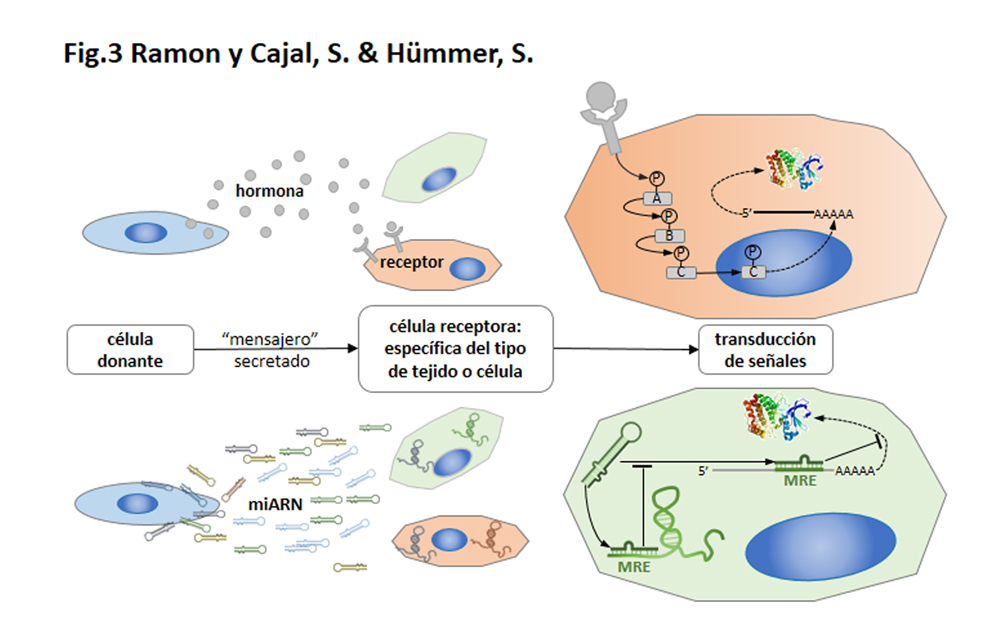
Este mecanismo propuesto a nivel intracelular, también puede extenderse a la comunicación entre diferentes células. Los miRNAs no solo actúan en las células en las que se transcriben, sino que también pueden transferirse a diferentes células (nivel intercelular). En este sentido, la interacción entre miRNAs y lncRNAs sugiere muchos paralelismos con las interacciones hormona-receptor. Las hormonas son liberadas como factores solubles por la célula donante, interactúan selectivamente con los receptores de la célula objetivo y activan las cascadas de señalización que finalmente alteran el fenotipo de la célula receptora. Se ha demostrado que los miRNAs se liberan en el espacio extracelular, y los lncRNAs se expresan de una manera específica de órgano, tejido o tipo celular. Como se describió anteriormente, la abundancia de lncRNAs puede determinar en última instancia el efecto de los miRNAs en la expresión de genes codificantes de proteínas. Por lo tanto, como ocurre en el modelo de receptor de hormonas, la exposición al mismo miARN puede resultar en alteraciones específicas de tipo celular que dependen de la expresión de los ncARN.
En resumen, se considera que estamos ante un rompecabezas con miles de piezas, tanto de alteraciones genéticas codificantes como no codificantes, epigenéticas, bioquímicas, etc. Hay un gran desconocimiento todavía del papel de los RNA no codificantes, así como de la especificidad tisular de los mismos. En conclusión, con los actuales paradigmas de biología molecular y celular, no se puede explicar el enorme puzle de factores implicados en la señalización celular y la especificidad tisular y celular. El arsenal de long non coding RNA puede representar la llave para avanzar en el conocimiento de la comunicación celular y de la especificidad celular y bioquímica.
BIBLIOGRAFÍA
- Portin P. The birth and development of the DNA theory of inheritance: sixty years since the discovery of the structure of DNA. J Genet 2014;93(1):293-302.
- McGranahan N, Swanton C. Biological and therapeutic impact of intratumor heterogeneity in cancer evolution. Cancer Cell 2015;27(1):15-26.
- Chapman A, Fernandez del Ama L, Ferguson J, Kamarashev J, Wellbrock C, Hurlstone A. Heterogeneous tumor subpopulations cooperate to drive invasion. Cell Reports 2014;8(3):688-695.
- Cheung KJ, Ewald AJ. A collective route to metastasis: Seeding by tumor cell clusters. Science 2016;352(6282):167-169.
- Ramon Y Cajal S, Capdevila C, Hernandez-Losa J, et al. Cancer as an ecomolecular disease and a neoplastic consortium. Biochim Biophys Acta 2017;1868(2):484-499.
- Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation. Cell 2011;144(5):646-674.
- Maddipati R, Stanger BZ. Pancreatic Cancer Metastases Harbor Evidence of Polyclonality. Cancer Discov 2015;5(10):1086-1097.
- Berk AJ. Discovery of RNA splicing and genes in pieces. P Natl Acad Sci USA 2016;113(4):801-805.
- Ulitsky I, Shkumatava A, Jan CH, Sive H, Bartel DP. Conserved function of lincRNAs in vertebrate embryonic development despite rapid sequence evolution. Cell 2011;147(7):1537-1550.
- Chan JJ, Tay Y. Noncoding RNA:RNA Regulatory networks in cancer. Int J Mol Sci 2018;19(5):1310.
- Guttman M, Amit I, Garber M, et al. Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals. Nature 2009;458(7235):223-227.
- Ponting CP, Oliver PL, Reik W. Evolution and functions of long noncoding RNAs. Cell 2009;136(4):629-641.
- Housman G, Ulitsky I. Methods for distinguishing between protein-coding and long noncoding RNAs and the elusive biological purpose of translation of long noncoding RNAs. Biochim Biophys Acta 2016;1859(1):31-40.
- Quinn JJ, Chang HY. Unique features of long non-coding RNA biogenesis and function. Nat Rev Genet 2016;17(1):47-62.
- Hon CC, Ramilowski JA, Harshbarger J, et al. An atlas of human long non-coding RNAs with accurate 5′ ends. Nature 2017;543(7644):199-204.
- Lagarde J, Uszczynska-Ratajczak B, Carbonell S, et al. High-throughput annotation of full-length long noncoding RNAs with capture long-read sequencing. Nat Genet 2017;49(12):1731-1740.
- Fok ET, Scholefield J, Fanucchi S, Mhlanga MM. The emerging molecular biology toolbox for the study of long noncoding RNA biology. Epigenomics-NY 2017;9(10):1317-1327.
- Cabili MN, Trapnell C, Goff L, et al. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Gene Dev 2011;25(18):1915-1927.
- Lv D, Wang X, Dong J, et al. Systematic characterization of lncRNAs’ cell-to-cell expression heterogeneity in glioblastoma cells. Oncotarget 2016;7(14):18403-18414.
- Chen J, Wang R, Zhang K, Chen LB. Long non-coding RNAs in non-small cell lung cancer as biomarkers and therapeutic targets. J Cell Mol Med 2014;18(12):2425-2436.
- Ulitsky I. Evolution to the rescue: using comparative genomics to understand long non-coding RNAs. Nat Rev Genet 2016;17(10):601-614.
- Goyal A, Myacheva K, Gross M, Klingenberg M, Duran Arque B, Diederichs S. Challenges of CRISPR/Cas9 applications for long non-coding RNA genes. Nucleic Acids Res 2017;45(3):e12.
- Aune TM, Crooke PS, 3rd, Patrick AE, Tossberg JT, Olsen NJ, Spurlock CF, 3rd. Expression of long non-coding RNAs in autoimmunity and linkage to enhancer function and autoimmune disease risk genetic variants. J Autoimmun 2017;81:99-109.
- Bhan A, Soleimani M, Mandal SS. Long noncoding RNA and cancer: A new paradigm. Cancer Res 2017;77(15):3965-3981.
- Viereck J, Thum T. Circulating noncoding RNAs as biomarkers of cardiovascular disease and injury. Circ Res 2017;120(2):381-399.
- Wan P, Su W, Zhuo Y. The role of long noncoding RNAs in neurodegenerative diseases. Mol Neurobiol 2017;54(3):2012-2021.
- Bussemakers MJ, van Bokhoven A, Verhaegh GW, et al. DD3: a new prostate-specific gene, highly overexpressed in prostate cancer. Cancer Res 1999;59(23):5975-5979.
- Wei JT, Feng Z, Partin AW, et al. Can urinary PCA3 supplement PSA in the early detection of prostate cancer? J Clin Oncol 2014;32(36):4066-4072.
- Gupta RA, Shah N, Wang KC, et al. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature 2010;464(7291):1071-1076.
- Bhan A, Mandal SS. LncRNA HOTAIR: A master regulator of chromatin dynamics and cancer. Biochim Biophys Acta 2015;1856(1):151-164.
- Huarte M. The emerging role of lncRNAs in cancer. Nat Med 2015;21(11):1253-1261.
- Freedman ML, Monteiro AN, Gayther SA, et al. Principles for the post-GWAS functional characterization of cancer risk loci. Nat Genet 2011;43(6):513-518.
- Li Y, Egranov SD, Yang L, Lin C. Molecular mechanisms of long noncoding RNAs-mediated cancer metastasis. Gene Chromosome Canc 2019;58(4):200-207.
- Ling H, Spizzo R, Atlasi Y, et al. CCAT2, a novel noncoding RNA mapping to 8q24, underlies metastatic progression and chromosomal instability in colon cancer. Genome Res 2013;23(9):1446-1461.
- Zhang ZZ, Shen ZY, Shen YY, et al. HOTAIR Long Noncoding RNA Promotes Gastric Cancer Metastasis through Suppression of Poly r(C)-Binding Protein (PCBP) 1. Mol Cancer Ther 2015;14(5):1162-1170.
- Nakagawa T, Endo H, Yokoyama M, et al. Large noncoding RNA HOTAIR enhances aggressive biological behavior and is associated with short disease-free survival in human non-small cell lung cancer. Biochem Bioph Res Co 2013;436(2):319-324.
- Prensner JR, Chinnaiyan AM. The emergence of lncRNAs in cancer biology. Cancer Discov 2011;1(5):391-407.
- Karreth FA, Tay Y, Perna D, et al. In vivo identification of tumor – suppressive PTEN ceRNAs in an oncogenic BRAF-induced mouse model of melanoma. Cell 2011;147(2):382-395.
- Salmena L, Poliseno L, Tay Y, Kats L, Pandolfi PP. A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language? Cell 2011;146(3):353-358.
- Tay Y, Rinn J, Pandolfi PP. The multilayered complexity of ceRNA crosstalk and competition. Nature 2014;505(7483):344-352.
- Ulitsky I. Interactions between short and long noncoding RNAs. FEBS Lett 2018;592(17):2874-2883.
- Thomson DW, Dinger ME. Endogenous microRNA sponges: evidence and controversy. Nat Rev Gene 2016;17(5):272-283.
- Zhu HY, Bai WD, Ye XM, Yang AG, Jia LT. Long non-coding RNA UCA1 desensitizes breast cancer cells to trastuzumab by impeding miR-18a repression of Yes-associated protein 1. Biochem Bioph Res Co 2018;496(4):1308-1313.
- Xie P, Cao H, Li Y, Wang J, Cui Z. Knockdown of lncRNA CCAT2 inhibits endometrial cancer cells growth and metastasis via sponging miR-216b. Cancer Biomark 2017;21(1):123-133.
- Cui M, You L, Ren X, Zhao W, Liao Q, Zhao Y. Long non-coding RNA PVT1 and cancer. Biochem Bioph Res Co 2016;471(1):10-14.
- Cheng DL, Xiang YY, Ji LJ, Lu XJ. Competing endogenous RNA interplay in cancer: mechanism, methodology, and perspectives. Tumor Biol 2015;36(2):479-488.
- Wang Y, Hou J, He D, et al. The Emerging Function and Mechanism of ceRNAs in Cancer. Trends Genet 2016;32(4):211-224.
- Ergun S, Oztuzcu S. Oncocers: ceRNA-mediated cross-talk by sponging miRNAs in oncogenic pathways. Tumor Biol 2015;36(5):3129-3136.
- Karreth FA, Reschke M, Ruocco A, et al. The BRAF pseudogene functions as a competitive endogenous RNA and induces lymphoma in vivo. Cell 2015;161(2):319-332.
- Liz J, Esteller M. lncRNAs and microRNAs with a role in cancer development. Biochim Biophys Acta 2016;1859(1):169-176.
Apoyo financiero: SRYC admite el apoyo del Fondo de Investigaciones Sanitarias (P1170185, PI17/000564), Generalitat de Catalunya (AGAUR, 2017 SGR 9015), y CIBERONC 2017 (CB16/12/00363).
DECLARACIÓN DE TRANSPARENCIA
El autor/a de este artículo declara no tener ningún tipo de conflicto de intereses respecto a lo expuesto en la presente revisión.
ranm tv
Santiago Ramón y Cajal
Real Academia Nacional de Medicina de España
C/ Arrieta, 12 · 28013 Madrid
Tlf.: +34 91 159 47 34 | sramon@vhebron.net
Año 2018 · número 135 (03) · páginas 230 a 236
Enviado*: 16.10.18
Revisado: 27.10.18
Aceptado: 22.11.18
* Fecha de lectura en la RANM


