Resumen
Mi objetivo es analizar desde una aproximación crítica los niveles de evidencia y los grados de recomendación más aceptados en la actualidad por la comunidad científica médica, evaluando críticamente el concepto de verdad en medicina y su incertidumbre. Tras ello, se analizará el papel de la radiómica y los biomarcadores de imagen como la aproximación científica de la imagen a la Medicina Personalizada, poniendo en valor los estudios observacionales con datos digitales in silico con biobancos de imágenes médicas para generar evidencia en el mundo real (Real World Data -RWD, Real World Evidence -RWE). Finalmente, se establecerá una propuesta metodológica para obtener la mejor inferencia de los datos observacionales asistenciales.
Esta propuesta incide en el desarrollo de estudios clínicos observacionales in-silico, obtenidos de forma disociada a los pacientes. Estos estudios utilizan repositorios bien estructurados para poder generar resultados de alta calidad para el sistema sanitario. Estos datos del mundo real (RWD) permiten, al ser analizados como ensayos clínicos experimentales con cohortes virtuales, obtener inferencias con un alto grado de evidencia (RWE) si se controla la calidad de los datos, las variables de confusión y los sesgos metodológicos. Esta evidencia puede ser más reproducible y fiable, más generalizada y universal, y sobre aspectos también más relevantes para los sistemas sanitarios.
La utilización de una gran cantidad de información (macrodatos, RWD, informes estructurados) y la capacidad del procesamiento computacional (radiómica y biomarcadores de imagen, Deep Learning) minimizará el problema del submuestreo, la heterogeneidad de la muestra y la variabilidad asociada a la falta de una estandarización de las imágenes. La Inteligencia Artificial ayudará en la evaluación crítica de los niveles de evidencia y los grados de recomendación, la verdad y su incertidumbre, la implementación de estudios observacionales in-silico con biobancos de imágenes médicas, y la obtención de la mejor inferencia con estos diseños.Abstract
My objective is to analyze, from a critical approach, the levels of evidence and the recommendation degrees most actually accepted by the medical scientific community, with a critical evaluation of the concept of truth and its uncertainty. Then, I will analyze the role of radiomics and imaging biomarkers as the scientific approximation of Imaging to Personalized Medicine. The value of observational in-silico studies with imaging biobanks to generate evidence in the real world (Real World Data – RWD, Real World Evidence – RWE) will be highlighted. Finally, a methodological proposal will be established to obtain the best inference of the observational assistance data.
This proposal relates to the development of observational clinical studies with digital data (in-silico), obtained in a dissociated form from the patients. These studies use well-structured repositories to generate high quality data for the health system. The Real-world data (RWD) allows, when analyzed as experimental clinical studies with virtual cohorts, to obtain inferences with a high degree of evidence (RWE) once data quality, confounding variables, and biases have been controlled. This evidence can be more reproducible and reliable, more generalized and universal, and also more relevant to health systems than standard prospective approaches.
The use of a large amount of information (data, RWD, structured reports) and the capacity of computational processing (radiomics, image biomarkers, Deep Learning) will minimize the problem of subsampling, sample heterogeneity, and data variability associated with the lack of standardization of images. Artificial Intelligence will assist in the critical evaluation of evidence levels and degrees of recommendation, truth and uncertainty, implementation of observational in-silico studies with imaging biobanks and obtaining the best inference with these designs.Palabras clave: Biomarcadores de Imagen; Biobancos de Imágenes; Macrodatos; Estudios Retrospectivos Observacionales; Inteligencia Artificial.
Keywords: Imaging Biomarkers; Imaging Biobanks; Big Data; Retrospective Observational Studies; Artificial Intelligence.
INTRODUCCIÓN
Existen en Medicina muchas incertidumbres con respecto al mejor método científico que puede emplearse para generar evidencias asistenciales y al papel de la imagen médica en esta inferencia de relaciones causa-efecto.
El objetivo de este trabajo es analizar desde una aproximación crítica los niveles de evidencia y los grados de recomendación aceptados en la actualidad por la comunidad científica médica, evaluar críticamente el concepto de verdad en medicina y valorar su incertidumbre; reconocer el papel de la radiómica y los biomarcadores de imagen como aproximación científica de la imagen a la Medicina Personalizada; poner en valor los estudios observacionales in-silico con biobancos de imágenes médicas para generar evidencia a partir de datos provenientes del mundo real (Real World Data -RWD, Real World Evidence -RWE) frente a los datos investigación (Research Controlled Data, RCD); y establecer una propuesta metodológica para obtener la mejor inferencia de los datos observacionales asistenciales.
Para ello tomaremos como ejemplo final el proyecto PRIMAGE (Research and Innovation Action -RIA del programa Horizon 2020) del que somos coordinadores. Este proyecto realiza un análisis avanzado de eficiencia in-silico (computacional y multiescala) sobre un repositorio de macrodatos digitales (base de datos centralizada) para generar inferencias clínicas como modelos estimativos basados en estadística multivariante e inteligencia artificial para la ayuda a la toma de decisión (Clinical Decision Support System -CDSS) en cáncer pediátrico.
NIVELES DE EVIDENCIA Y DISEÑO DE ESTUDIOS CIENTÍFICOS
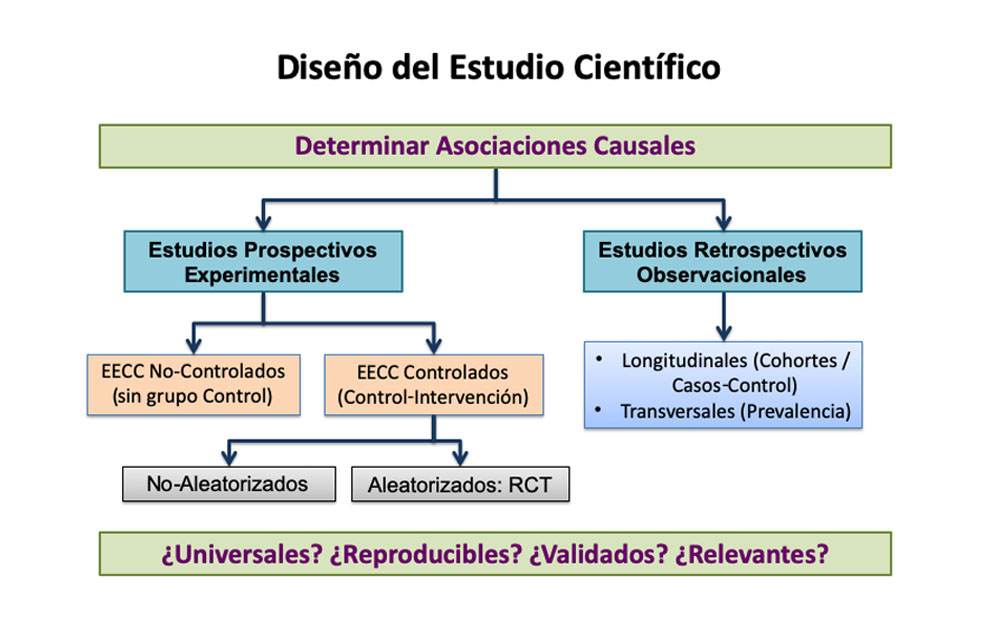
Las recomendaciones para establecer el valor de la evidencia y la potencia de la recomendación no están estandarizadas, aunque todas las clasificaciones existentes comparten propuestas muy similares. Un sistema sencillo y eficaz para determinar los niveles de evidencia, siendo unos de los más utilizados, se basa en las recomendaciones establecidas por el grupo de trabajo GRADE (Grading of Recommendations Assessment, Development and Evaluation) (1). En ellas se define el grado de conocimiento con el que podemos afirmar la validez de un contenido, es decir, si es verdadero; y su grado de incertidumbre, o sea, las dudas sobre la afirmación. Este sistema emplea tres categorías de evidencia, ajustadas a dos grados de recomendación. Como evidencia, clasifica como Alta (A) a los metanálisis, las revisiones sistemáticas y las conclusiones de ensayos clínicos múltiples y randomizados de alta calidad, estimando que sus términos no se modificarán con las publicaciones de nuevos estudios. Como evidencia Moderada (B) incluye las conclusiones de un ensayo clínico randomizado y los análisis de estudios múltiples no randomizados. Para esta evidencia de nivel B es posible que la publicación de nuevos estudios cambie la confianza en los beneficios y riesgos estimados. El nivel menor de evidencia es el Bajo (C), obtenido con el análisis de estudios observacionales retrospectivos desde los registros de datos, y también de los estudios con series pequeñas o no-consecutivas. Sus estimaciones se consideran que son inciertas. No se considera como evidencia sostenible la obtenida de casos individuales, editoriales ni opinión de expertos. Vemos que en este sistema las conclusiones de estudios observacionales retrospectivos se consideran como Evidencia Baja. Para estos niveles de evidencia (A, B, C) se ajustan los dos grados de recomendación: muy necesaria o sólida (grado 1) cuando la afirmación está justificada por una sociedad, asociándose a “debe hacerse”; y neutra o débil (grado 2) cuando una sociedad sugiere su uso como “puede hacerse” (1).
Como vemos, los estudios observacionales retrospectivos no se consideran válidos para generar evidencia de nivel alto, ni siquiera moderado, cuando se pretende determinar la existencia de asociaciones causales entre exposiciones o presencia de determinadas características, como los atributos radiómicos y los parámetros dinámicos obtenidos de las imágenes, y las enfermedades o sus fenotipos de comportamiento concreto o expresión diferencial. Y se necesitan estos niveles altos de evidencia, asociados en general a metanálisis y revisiones sistemáticas de estudios prospectivos experimentales, sin o con grupo control, sin o con randomización, para asumir que la correlación observada implica la existencia de una relación de causalidad (Figura 1). Son precisamente los resultados de múltiples estudios prospectivos controlados y randomizados (Randomized Clinical Trials, RCT) los que presentan un nivel de evidencia mayor. Pero, cabe preguntarse si estos estudios RCT generan resultados universales, reproducibles, válidos en la práctica habitual y relevantes en el manejo práctico de los pacientes.
LA CRISIS DE LA RELEVANCIA
La crisis de la validez estadística de los estudios publicados hasta 2005, señalada por Ioannidis (2), se solucionó en gran parte con un diseño metodológico más exhaustivo. Así, se indicaba que la probabilidad de que un resultado de investigación sea cierto depende de la potencia estadística del estudio, el control de los sesgos, el número de estudios diferentes con resultados similares, y la relación entre verdadero/falso. Un estudio es menos probable que sea cierto si la muestra es pequeña, los efectos de tamaño son pequeños, las variables son muchas, y la metodología es flexible (3). Estos problemas han llevado a implementar métodos estadísticos más rigurosos, con mejores aproximaciones al tamaño muestral necesario, minimizando la tasa de resultados falsos positivos (false discovery rates, FDR) y validando con series diferentes, preferentemente independientes (4). Parece pues imposible e inalcanzable conocer con exactitud la verdad absoluta en un estudio clínico. Sin embargo, una mayor potencia estadística con control del tamaño muestral y una ausencia de sesgos relevantes han mejorado la calidad de la inferencia causal en los estudios científicos en medicina. Además, pese a la existencia de diferencias estadísticas significativas, se ha hecho crítica la existencia de un impacto relevante en la práctica y el conocimiento médico para asumir un resultado como significativo.
A pesar de haber ajustado en gran medida estas consideraciones estadísticas, existe otro factor muy limitante para poder aplicar con seguridad los resultados de la investigación médica. Este otro aspecto se conoce como la crisis de reproducibilidad (5) y expresa que más del 70% de los investigadores que publican en Nature han intentado sin éxito reproducir estudios de otros autores; e incluso hasta un 50% han sido incapaces de reproducir sus propios estudios. Las publicaciones sobre estudios de replicación positivos son escasas y muchas revistas son reacias a publicar estos resultados negativos (5).
Aunque los factores como la presión por publicar y las publicaciones parciales o selectivas están detrás de esta falta de calidad, existen otros más profundos que debemos considerar aquí. Esta crisis está claramente relacionada con la heterogeneidad de las muestras analizadas. Cualquier población que se seleccione no es necesariamente representativa de la globalidad. Así, es preciso garantizar con la mayor seguridad posible que la muestra analizada y estudiada representa adecuadamente a la población general, es decir, al universo de posibilidades. Para ello debemos controlar tanto las variables relacionadas con la selección de los sujetos como la metodología de validación de los resultados obtenidos en una muestra totalmente independiente y con condiciones de observación diferentes.
Ambas crisis, la de validez estadística y la de reproducibilidad, están pues relacionadas con la calidad de la muestra analizada, su capacidad para representar a la población general, la potencia estadística de las diferencias, y la relevancia clínica de esas diferencias.
LA INFERENCIA CAUSAL: RETOS DE LA IMAGEN MÉDICA
Pretender que la medicina basada en la evidencia obligatoriamente requiera de estudios comparativos prospectivos y aleatorizados conduce a que muchas decisiones clínicas carezcan de una información pertinente. Aunque se estipula que los estudios prospectivos tienen un mayor nivel de evidencia que los retrospectivos, estos no siempre son posibles de establecer y su calidad puede incluso ser muy cuestionada.
En este sentido, tanto los estudios prospectivos, tipo Ensayos Clínicos, como los retrospectivos, especialmente los realizados con casos del mundo real (RWD), presentan sus ventajas pero también sus inconvenientes (6). La principal ventaja de los estudios prospectivos experimentales es el control y la homogeneidad de la muestra, con criterios de inclusión y de exclusión bien definidos desde el principio. Entre sus inconvenientes se incluyen el tamaño limitado de la muestra analizada, la no representatividad por los criterios de inclusión usualmente muy estrictos, su alto coste económico y, consecuentemente, la evidencia limitada al grupo seleccionado (sesgo de submuestreo). Por otro lado, los estudios retrospectivos con datos clínicos extraídos de condiciones de práctica habitual (RWD) presentan las ventajas de representar a grandes grupos de población, incluyendo sujetos con otras comorbilidades como covariables, con una ventana de inclusión más amplia, un menor coste económico y una generación de evidencia más universal (RWE) (7). Los inconvenientes de construir evidencia con RWD se deben a la mayor heterogeneidad de los datos extraídos y la necesidad asociada de controlar la calidad de estos datos (8).
El desarrollo tecnológico asociado a la revolución digital en Medicina ha propiciado reevaluar el papel de los estudios retrospectivos observacionales, realizados en un entorno computacional in-silico sobre datos de los sistemas de salud obtenidos de registros de almacenamiento. Esta aproximación permite el análisis de una gran cantidad de información (macrodatos, Big Data), estructurada y agrupada con un concepto de cohortes virtuales, para poder así inferir asociaciones causales con la potencia estadística suficiente y libres de sesgos de relevancia (9).
Estos macrodatos empleados en los estudios observacionales retrospectivos in-silico (EORIS) se extraen de diversas fuentes desde las que se alimenta un repositorio centralizado de datos anonimizados con información relevante para poder extraer inferencias de causalidad con la mayor evidencia. Las principales fuentes de información representativas de RWD para construir EORIS son los registros de pacientes que han participado en Ensayos Clínicos (nacionales, internacionales), los registros de pacientes con enfermedades raras (p.ej. ORPHANET, el REEG de la enfermedad de Gaucher y el de SIOPEN con Neuroblastoma), los biobancos públicos (como la Red Nacional de Biobancos – RETICS), los datos gubernamentales sobre Sistemas de Información Radiológica (como el Informe Estructurado) y los existentes en las Historias Clínicas Informatizadas (Electronic Health Records EHR, Hospital Information Systems HIS) con sus repositorios de datos sanitarios. El principal objetivo de acudir a estas fuentes es recoger un volumen amplio (macrodatos), con la mayor completitud de información, adecuados para la hipótesis que se está analizando, organizados y estandarizados acorde a la práctica clínica habitual. Para que estos datos sean adecuados deben cribarse, asegurando su calidad y validez (data curation).
Una iniciativa importantísima para recoger datos de la práctica clínica habitual de forma que estén estandarizados, sean pertinentes, y permitan su explotación estadística es el Informe Estructurado (Figura 2). Estos documentos utilizan menús desplegables con preguntas y repuestas pactadas en un entorno multidisciplinario, con una base de datos asociada, y con capacidad de incorporar datos radiómicos y parámetros dinámicos extraídos de las imágenes como biomarcadores específicos del problema clínico para el que se han desarrollado, tanto en forma de valores como de imágenes paramétricas (10).

LA REALIDAD CLÍNICA Y SU INFERENCIA POR IMAGEN
Podemos consensuar que la verdad es el arte de evaluar críticamente el conocimiento establecido para entender con la mayor precisión los procesos biopatológicos y sus causalidades. El problema de aproximarnos con la imagen médica a inferir la verdad no está exento de múltiples fuentes de error, siendo pues multifactorial en sus sesgos. Así, la señal adquirida del vóxel en cada imagen es compleja, con múltiples componentes y propiedades, por lo que asumir que su magnitud representa una única variable es una simplificación excesiva. De este modo, un parénquima hepático de atenuación normal puede asociarse a esteatosis con sobrecarga de hierro simultánea ya que ambos factores modifican la señal de forma opuesta. La imagen tiene también covariables propias que introducen heterogeneidad a los datos, tales como las diferencias en adquisición de imágenes y en el procesamiento de datos. En comparación con otros ámbitos bioanalíticos, tales como los análisis de sangre, la imagen médica es la modalidad de análisis que conlleva una mayor complejidad y variabilidad en la obtención de la muestra por los diferentes equipos y técnicas con las que se pueden obtener.
Por otro lado, existe una incapacidad de cualquier estudio observacional para detectar el espectro completo de la heterogeneidad biológica, ya que siempre es mayor este espectro que el submuestreo de pacientes y sujetos estudiados. Además, los resultados y objetivos finales tienen una alta variabilidad temporal, tanto en su expresión biológica determinada como en la situación clínica específica.
Estas incertidumbres en los EORIS, cuando se construyen para generar evidencia diagnóstica con la imagen computacional, deben controlarse para poder inferir resultados con niveles altos de evidencia. Los métodos analíticos de cuantificación radiológica deben cumplir con los requisitos críticos de tener consistencia conceptual, validar el rendimiento técnico (precisión y exactitud), y ser sensibles al rango de efectos asociados a un tratamiento, tanto a sus eventos adversos como a los resultados clínicos relevantes. Sin embargo, como hemos comentado, existe una alta heterogeneidad en la calidad de la imagen debida a las diferencias tecnológicas implementadas por los proveedores y los diferentes protocolos utilizados en distintos centros.
Por ello, debemos reconocer que en la práctica clínica la estandarización de las imágenes obtenidas para adecuar el análisis de los biomarcadores de imagen no es factible de forma universal (diferentes vendedores, distintas actualizaciones). Tal vez los enfoques basados en Inteligencia Artificial, principalmente las redes neuronales con volucionales profundas mediante técnicas más conocidas como Deep Learning, sean una manera disruptiva de cambiar esta tendencia, al hacer posible que estas redes complejas aprendan a controlar la heterogeneidad inherente a los datos de imagen. Es muy probable que la Inteligencia Artificial sea capaz de extraer datos, atributos y parámetros de las imágenes médicas, y de integrarlos en agrupaciones no supervisadas, incluso en entornos con mucha incertidumbre y variabilidad, mediante el reconocimiento de estas variaciones y la incorporación al análisis de los metadatos asociados.
LA RADIÓMICA Y LOS BIOMARCADORES DE IMAGEN EN LA MEDICINA PERSONALIZADA
La imagen médica ha contribuido notablemente al desarrollo de la medicina personalizada. En un principio, las diversas modalidades de imagen han contribuido en la detección, caracterización y estadiaje de numerosas lesiones y enfermedades. Con su utilización masiva, gran parte de los pacientes se benefician de esta precisión a la hora de definir si padecen una enfermedad, la localización de las lesiones y la estimación de la mejor aproximación terapéutica. Sin embargo, y pese a la gran resolución espacial obtenida en las imágenes tomográficas (tales como RM, TC y PET), la información que estas pruebas generan no es completa y presenta algunas lagunas para poder tomar decisiones individualizadas en base a la evidencia actual. Así, tras detectar un glioma cerebral, se desconocen aspectos tan importantes como la extensión de la infiltración peritumoral, la afectación de los fascículos de sustancia blanca adyacentes, el nivel regional de oxígeno para calcular la sensibilidad a la radioterapia, los subgrupos celulares de comportamiento diferentes, la neoangiogénesis y su sensibilidad a la terapia antiangiogénica, y el pronóstico como estimación de supervivencia como información a priori para ensayos clínicos.
En un intento de dar respuesta a estas necesidades, y otras muchas relevantes a otras enfermedades, se ha desarrollado la imagen médica computacional con los biomarcadores de imagen (11). Esta ciencia estudia los tejidos a partir de sus imágenes en un intento de describir aquellos fenómenos considerados como relevantes para el paciente, con exactitud y veracidad. Se necesita conocimiento para analizar mediante procesos computacionales los cambios biológicos tisulares asociados a la enfermedad desde sus imágenes médicas y así poder detectar, clasificar y representar estas anomalías. Estos datos extraídos por análisis computacional in-silico se emplean para construir modelos descriptivos y predictivos en base a las correlaciones con la realidad biopatológica y los resultados clínicos finales. Si se establecen estas correlaciones, entonces se consideran como biomarcadores de imagen.
En general, se considera biomarcadores a todas aquellas características que puedan medirse de forma objetiva y que se comporten como indicadores subrogados de procesos biológicos normales, cambios patológicos o respuestas farmacéuticas. En nuestro caso, los biomarcadores de imagen son parámetros cuantitativos subrogados, extraídos computacionalmente de las imágenes médicas. Estos datos, obtenidos píxel-a-píxel, pueden representarse como imágenes paramétricas donde la distribución y magnitud del cambio permite el análisis de la localización y heterogeneidad espacial. Además, y dado que no se destruye la muestra cuando se adquieren las imágenes, estos cambios pueden analizarse en estudios longitudinales, pudiendo considerarse como resueltas también en el tiempo.
Desde las imágenes adquiridas, conociendo los parámetros que se quieren calcular y los conceptos que se quieren inferir, y tras preparar las imágenes a un marco de referencia común espacial (corregistro, escalado) y de señal (filtrado de ruido y normalización de la señal), se extraen los datos computaciones desde dos entornos principales (Figura 3). Por un lado, está el dominio de los atributos de la imagen, desde el que se computan texturas a partir del histograma de las intensidades (para extraer métricas como la curtosis, asimetría –skewness-, entropía, y energía), la distribución espacial de la señal (para obtener datos como las matrices GLCM -Gray Level Co-Occurrence Matrix- y GLRLM -Gray Level Run-Length Matrix-, filtros gradilla, fractales, y ondículas), y los descriptores morfológicos (entre los que podemos incluir aquellos de volumen, espesor, forma, bordes, vasos, y sus cambios temporales). Por el otro lado, se consideran los parámetros obtenidos del análisis de series dinámicas donde existen modificaciones de la señal, como por ejemplo los modelos de perfusión (heurístico, farmacocinético), difusión (monoexponencial, multiexponencial), los ratios de relajación (valores de T1 y T2 en RM) y el análisis espectral de absorción en TC. Todos estos atributos y parámetros, antes de correlacionarse como biomarcadores subrogados, deben reducirse para evitar redundancias e inestabilidades que impidan un análisis correcto y preciso de estas relaciones.
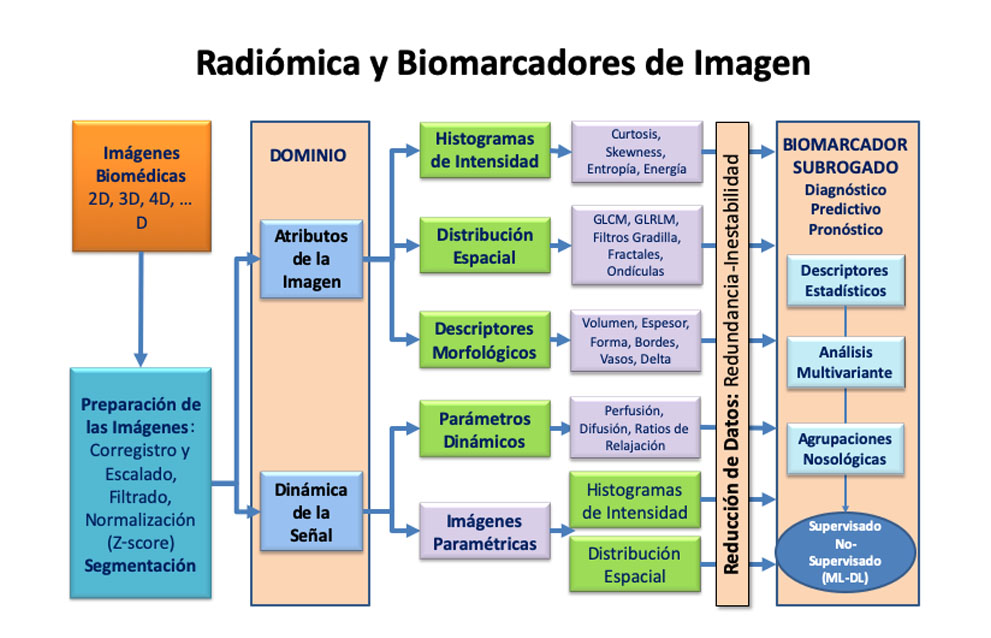
Para establecer la correlación de los datos computacionales con estos valores (diagnóstico de fenotipo, predictivo de tratamiento o pronóstico de resultado clínico) pueden emplearse tanto descriptores estadísticos clásicos, análisis multivariante y agrupaciones nosológicas. También pueden relacionarse mediante modelos de agrupamiento no supervisado empleando Inteligencia Artificial, con métodos basados en agrupamientos (clusterición) jerárquicos. Estos métodos pueden dar el resultado para todo el tumor o para sus hábitats o componentes principales. Estos hábitats expresan regiones de comportamiento similar obtenidos como firmas nosológicas multiparamétricas, bajo la premisa de que para cada evento biopatológico hay una firma nosológica que puede extraerse.
Así, y en líneas generales, los biomarcadores identificados se clasifican según su objetivo estimativo en (12):
- Diagnóstico/Fenotipado: aquellos que detectan o confirman la presencia de un proceso patológico, una enfermedad concreta o un subtipo de la enfermedad. Estas métricas son marcadores subrogados de agresividad, incluyendo tiempo de duplicación de volumen, invasión local y extensión a distancia. En este grupo están los biomarcadores de susceptibilidad, que son aquellos cuya presencia identifica el potencial para desarrollar una enfermedad.
- Predictivo: características que señalan a los individuos con más probabilidades de experimentar un efecto favorable (o desfavorable) con el tratamiento. Se consideran en esta categoría a los biomarcadores de respuesta, cuando muestran el efecto biológico generado por el tratamiento, y de seguridad, cuando indican la probabilidad, presencia o grado de toxicidad como efecto adverso al tratamiento. Estas métricas son marcadores subrogados relacionados con el tratamiento, incluyen aspectos como los ratios coste/beneficio (coste material), riesgo/beneficio (toxicidad evitable) y oportunidad/beneficio (tiempo a efecto).
- Pronóstico: valores que determinan la probabilidad o el tiempo a un evento clínico, a una recurrencia o a la progresión de la enfermedad, y la supervivencia del paciente. Estas métricas son marcadores subrogados relacionados con la respuesta clínica objetiva.
Pero antes de considerarlos como biomarcadores, los datos de imagen computacional extraídos como descriptores simples o en asociación (firmas tisulares), deben validarse. El proceso de validación se debe establecer en dos niveles principales:
- Por un lado, está la Validación Técnica que analiza la variabilidad en su proceso de obtención. Para ello se debe controlar la posible redundancia de datos muy correlacionados y eliminar además los que no sean reproducibles por inestabilidad en la extracción del atributo. Se debe controlar también su precisión y estabilidad con el coeficiente de correlación intraclase, aplicado a la repetición de experimentos sin cambios (repetitividad para los mismos equipos con estudios tipo test-retest) y a la repetición con cambios (reproducibilidad en otros equipos con datos independientes). Otro aspecto fundamental en esta validación técnica es el estudio de la exactitud de las medidas mediante el análisis de su asociación con la verdad, considerada como la realidad de los dispositivos tipo fantomas o de la referencia patológica. Esta validación en la exactitud permite además establecer calibraciones para mejorar la relación de un método concreto de obtener el biomarcador con la verdad.
- Por otro lado, tenemos la Validación Clínica que analiza la relevancia de la asociación en el entorno asistencial, es decir, su eficiencia práctica. Así, la fiabilidad se establece con la relación existente entre el biomarcador y los resultados clínicos definidos como objetivos en la prueba de concepto, y la de impacto determina si se producen cambios en la práctica clínica asociados a la presencia de estas nuevas aproximaciones.
BIOBANCOS DE IMÁGENES MÉDICAS Y EVIDENCIA EN EL MUNDO REAL
Los biobancos se consideran como depósitos organizados de muestras biológicas. Estas colecciones son fundamentales para la investigación clínica y la innovación en la medicina personalizada. Su estructura dispone de controles de calidad en la colección de las muestras, estandarización para su manipulación y protocolos legales para la protección de sus datos. Dada la necesidad de correlacionar sus datos para inferir relaciones más fiables, los biobancos pueden estar relacionados con otros repositorios de datos manteniendo una trazabilidad de los sujetos.
Los biobancos de imágenes son repositorios estructurados de las imágenes digitales asociadas con episodios clínicos y sus metadatos (13). Estos biobancos virtuales in-silico se crean para avanzar en el estudio de las enfermedades raras, la identificación de biomarcadores subrogados tempranos, y el desarrollo de las imágenes computacionales en los estudios EORIS. Un ejemplo de biobanco es el nodo valenciano de Euro-BioImaging (ESFRI europea). Este biobanco se construye para evaluar en abierto, dando soporte a los investigadores que soliciten acceso a través de convocatorias abiertas, el impacto de los nuevos biomarcadores en el diagnóstico temprano de la enfermedad, su fenotipado, gradación, terapias dirigidas, evaluación de la respuesta al tratamiento y sus posibles efectos adversos. Este Nodo ofrece, además, a través de sus relaciones consorciadas, el acceso a las herramientas de procesamiento de imágenes, extracción de datos radiómicos y de biomarcadores de imagen, simulación biomédica, inteligencia artificial y bioinformática, minería de datos, y desarrollo ad-hoc de productos y recursos computacionales de altas prestaciones.
Los principales servicios prestados por un biobanco de imágenes como el nodo valenciano de Euro-BioImaging, incluyen gestionar las imágenes (aceptación, etiquetado, formatos, modalidades, calidad, metadatos, enlace a datos clínico-biológicos externos), proveer de datos (comité de acceso, comité ético, procesos de consulta, anonimización, recuperación de información externa relacionada), generar y validar hipótesis diagnósticas, predictoras y pronósticas con macrodatos y biomarcadores de imagen (precisión, exactitud, intervalos de confianza) y, finalmente, establecer inferencias entre los fenotipos computacionales y la evolución clínica.
METODOLOGÍA PARA INFERIR EVIDENCIA DE DATOS OBSERVACIONALES ASISTENCIALES
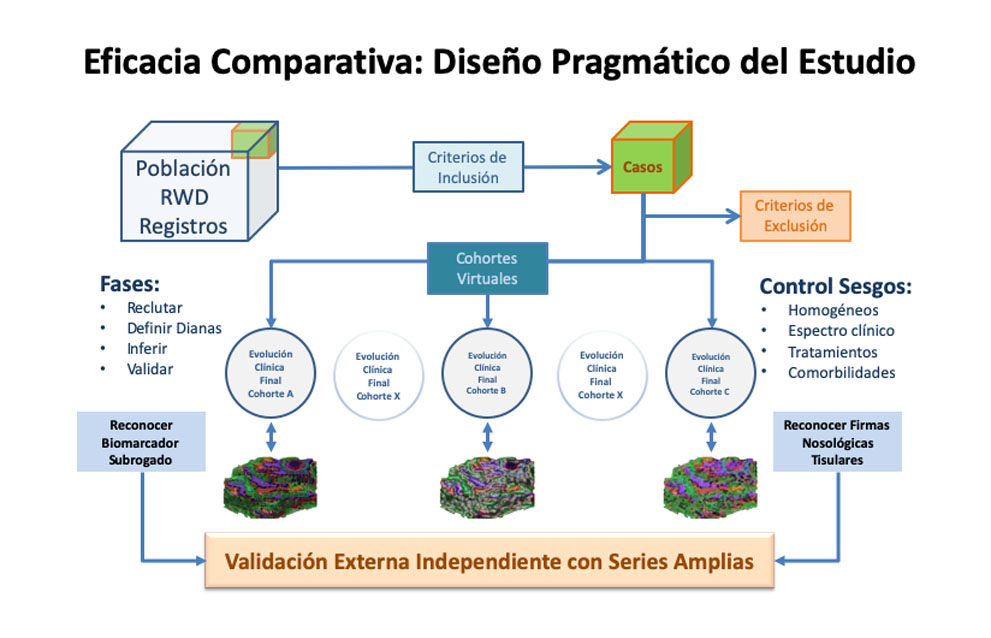
Cuando se recogen datos del mundo real (RWD) para emular estudios experimentales prospectivos (tipo RCT), deben definirse y establecerse los mismos parámetros y metodologías. De esta forma ambos estudios se aproximan para obtener un nivel alto de evidencia para inferir causalidades en los resultados (RWE) (Figura 4). Entre los principales aspectos a definir antes de comenzar a recoger datos retrospectivos debemos considerar (9,14):
- Criterios de inclusión en el reclutamiento de pacientes: ¿se ha basado en la presencia de síntomas, resultados de pruebas anteriores, existencia de estudios índice, poder padecer una enfermedad o tenerla confirmada con un estándar de referencia?
- Selección de la muestra de participantes: ¿se recogieron los casos de forma consecutiva?, ¿se explica cómo se seleccionaron los participantes?, ¿están definidos los criterios de exclusión?
- Control de las variables de confusión: ¿se han considerado como covariables las características sistemáticas de confusión que puedan influir en el resultado final, como las diferencias epidemiológicas, de fenotipo, gradación, estadio, intervalos temporales y tratamientos?, ¿se han considerado las diferencias metodológicas que puedan inducir a error?
- Recopilación de datos: ¿se planificó cómo recoger la información antes del muestreo? ¿Puede simularse la recogida de datos como en un ensayo clínico?
- Patrón de referencia: ¿se ha justificado y descrito su uso?, ¿está referenciado?
- Observadores: ¿se ha descrito el número, capacitación y experiencia de las personas que realizan las pruebas, obtienen los biomarcadores e interpretan los resultados de las pruebas índice y del patrón de referencia?
- Estudio ciego: ¿se ha descrito si los lectores de las pruebas y del patrón de referencia conocían los resultados de otras pruebas o el estado clínico final del paciente?
- Especificaciones técnicas en Material y Métodos: ¿se describe cómo y cuándo se obtuvieron las imágenes?, ¿están definidos los atributos y marcadores de imagen derivados?
- Decisiones internas: ¿se han definido las unidades empleadas, los puntos de corte y las categorías de los resultados? ¿se han descrito todos los tratamientos?
- Control de sesgos: ¿se han descrito los tiempos entre procesos en la práctica clínica habitual?, ¿se han valorado los criterios de asignación de tratamiento empleados y sus posibles desviaciones?
Así, para que un estudio EORIS pueda emular un ensayo clínico (Target Trial) y obtener modelos estimativos objetivos para evaluar inferencias con los datos de imagen computacional, los biomarcadores de imagen (datos radiómicos y parámetros dinámicos) se deben considerar como covariables “no-expuestas” ya que no se tuvieron en cuenta en la toma de decisiones prácticas. De esta forma pueden inferirse resultados explicativos con cohortes virtuales de enfermedades para descubrir biomarcadores (guiadas por la enfermedad) o cohortes agrupadas por el resultado de los biomarcadores y así estimar fenotipos y respuestas (guiadas por la imagen).
EL EJEMPLO DE PRIMAGE (RIA H2020, PROYECTO 826494)
Los biobancos centralizados son repositorios que constituyen una plataforma integrada y estructurada. Su objetivo principal es proporcionar acceso a los datos y servicios de procesamiento de imágenes, para estudios clínicos observacionales con datos disociados, y así poder emular ensayos clínicos observacionales in-silico.
El acceso abierto a sus infraestructuras debe cumplir con la normativa de protección de datos y requiere de una estructura definida. Entre los aspectos más importantes de su organización cabe destacar la incorporación de nuevos datos, una ontología común estandarizada, los rangos y espectros de enfermedades, su dependencia organizativa, la estratificación de la afectación, la asociación con otros datos clínicos y biológicos como covariables, los criterios necesarios previos a la solicitud de acceso, y la existencia de comités de acceso y de dirección de esta infraestructura (Figura 4).
Su marco computacional intensivo proporciona a los investigadores, como valor añadido, diversas herramientas, datos metodológicos y experiencia previa para mejorar la investigación con el uso de fenotipos computacionales que permitan generar una inferencia casual de calidad para una mejor atención médica a los pacientes. Su objetivo final es ofrecer en abierto un sistema de ayuda a la toma de decisiones para aquellos niños con diagnóstico inicial de neuroblastoma y gliomas difusos infiltrativos de puente a través de la imagen computacional.
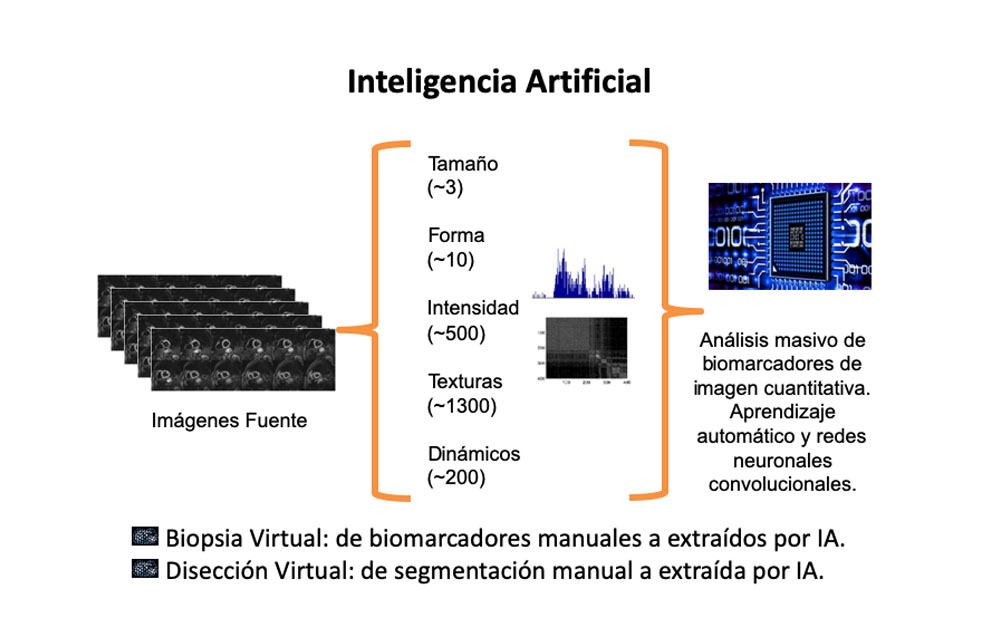
Para ofrecer esta ayuda en condiciones de reproducibilidad y rapidez, se utilizarán datos del mundo real (RWD) e inteligencia artificial. Las soluciones de Deep Learning generarán datos de calidad en el entorno de la Disección Virtual (aprendiendo desde la segmentación manual a la automática cómo separar órganos y lesiones para su análisis individual) y de la Biopsia Virtual (como extraer automáticamente de forma robusta a la falta inherente de estandarización en imagen médica los diversos biomarcadores de imagen, uni- y multi-variantes) (Figura 5).
CONCLUSIONES
Los ensayos clínicos observacionales con datos digitales in-silico, obtenidos de forma disociada a los pacientes, deben utilizar repositorios bien estructurados para así generar resultados de alta calidad para el sistema sanitario. La utilización de una gran cantidad de información (macrodatos, RWD, informes estructurados) y la capacidad del procesamiento computacional (radiómica y biomarcadores de imagen, Deep Learning) minimizará el problema del submuestreo, la heterogeneidad de la muestra y la variabilidad asociada a la falta de una estandarización de las imágenes.
Estos datos del mundo real (RWD) permiten, al ser analizados como ensayos clínicos experimentales con cohortes virtuales, obtener inferencias con un alto grado de evidencia (RWE) si se controla la calidad de los datos, las variables de confusión y los sesgos metodológicos. Esta evidencia puede ser más reproducible y fiable, más generalizada y universal, y sobre aspectos también más relevantes para los sistemas sanitarios.
La Inteligencia Artificial nos ayudará en la evaluación crítica de los niveles de evidencia y los grados de recomendación, la verdad y su incertidumbre, la implementación de estudios observacionales in-silico con biobancos de imágenes médicas, y la obtención de la mejor inferencia de estos diseños.
AGRADECIMIENTOS
Muy especialmente al personal del Grupo de Investigación Biomédica en Imagen (GIBI230) y de la Plataforma de Radiología Experimental y Biomarcadores de Imagen (PREBI) en el Instituto de Investigación Sanitaria La Fe por sus aportaciones en la concepción y diseño de esta metodología.
BIBLIOGRAFÍA
- Guyatt GH, Oxman AD, Vist GE, et al. GRADE: an emerging consensus on rating quality of evidence and strength of recommendations. BMJ. 2008;336(7650):924-926.
- Ioannidis JP. Why most published research findings are false. PLoS Med. 2005; 2(8):e124.
- An G. The Crisis of reproducibility, the denominator problem and the scientific role of multi-scale modeling. Bull Math Biol. 2018; 80(12):3071-3080.
- Foster KR, Skufca J. The Problem of false discovery: many scientific results can’t be replicated, leading to serious questions about what’s true and false in the world of research. IEEE Pulse. 2016; 7(2):37-40.
- Baker M. 1,500 scientists lift the lid on reproducibility. Nature. 2016; 533(7604):452-454.
- Booth CM, Karim S, Mackillop WJ. Real-world data: towards achieving the achievable in cancer care. Nat Rev Clin Oncol. 2019. doi: 10.1038/s41571-019-0167-7.
- Sherman RE, Anderson SA, Dal Pan GJ, et al. Real-World Evidence – What is it and what can it tell us? N Engl J Med. 2016; 375(23):2293-2297.
- Dubois RW. Is the real-world evidence or hypothesis: a tale of two retrospective studies. J Comp Eff Res. 2015; 4(3):199-201
- Von Elm E, Altman DG, Egger M, et al. The Strengthening the Reporting of Observational Studies in Epidemiology (STROBE) statement: guidelines for reporting observational studies. Ann Intern Med. 2007; 147(8):573-577.
- Martí-Bonmatí L, Ruiz-Martínez E, Ten A, Alberich-Bayarri A. Cómo integrar la información cuantitativa en el informe radiológico del paciente oncológico. Radiología. 2018; 60(1):43-52.
- Martí Bonmatí L, Alberich-Bayarri A, García-Martí G, et al. Biomarcadores de imagen, imagen cuantitativa y bioingeniería. Radiología. 2012; 54(3):269-278.
- https://www.fda.gov/Drugs/DevelopmentApprovalProcess/DrugDevelopmentToolsQualificationProgram/BiomarkerQualificationProgram/ucm535922.htm
- European Society of Radiology. ESR Position paper on imaging biobanks. Insights Imaging. 2015; 6(4):403-410.
- Hernán MA, Robins JM. Using Big Data to emulate a target trial when a randomized trial is not available. Am J Epidemiol. 2016; 183(8):758-764.
DECLARACIÓN DE TRANSPARENCIA
El autor/a de este artículo declara no tener ningún tipo de conflicto de intereses respecto a lo expuesto en la presente revisión.
ranm tv
Luis Martí-Bonmatí
Real Academia Nacional de Medicina de España
C/ Arrieta, 12 · 28013 Madrid
Tlf.: +34 91 159 47 34 | luis.marti@uv.es
Año 2019 · número 136 (01) · páginas 34 a 42
Enviado*: 12.03.19
Revisado: 19.03.19
Aceptado: 22.04.19
* Fecha de lectura en la RANM


