Resumen
Se estudia primero la morfología y estructura del virus para, en una segunda parte, actualizar las técnicas diagnósticas de laboratorio, los problemas que plantean y las incertidumbres actuales de muchas de ellas.
Abstract
The virus morphology and structure are studied first, in a second part to update the diagnostic laboratory techniques, the problems they pose and the current uncertainties of many of them.
Palabras clave: Coronavirus; SARS-CoV-2; Técnicas de diagnóstico de laboratorio; Problemas e incertidumbres del SARS-CoV-2.
Keywords: Coronavirus; Laboratory diagnostic techniques; SARS-CoV-2; SARS-CoV-2 issues and uncertainties.
Introducción
La enfermedad del Covid-19 está causada por el coronavirus SAR-CoV-2. Ha sido declarada pandemia por la Organización Mundial de la Salud y causado cientos de muertos (Más de 450.000 al día de hoy 19/6/2020), afectando a más de 195 países del mundo. La falta de inmunidad previa para el virus Covid-19 es la que ha condicionado esta rápida progresión de la enfermedad.
Los pulmones son el órgano diana en la infección, pero pueden también afectarse otros órganos, con muerte de los pacientes por fallo multiorgánico.
En este trabajo analizamos el proceso microbiológico que conlleva al diagnóstico precoz y tardío, así como los problemas que plantea.
De forma esquemática planteamos nuestro trabajo en cuatro apartados:
- Morfología, estructura y replicación del virus.
- Técnicas de laboratorio para el diagnóstico.
- Planteamiento de dudas, problemas y, sobre todo, presencia de incertidumbres en la infección por SARS.CoV-2.
- ¿Qué es lo que podemos y debemos buscar para solucionar los problemas planteados?
Morfología, estructura y replicación del virus
Los coronavirus son miembros de la subfamilia Orthocoronavirinae dentro de la familia Coronaviridae (orden Nidovirales). De acuerdo a su estructura genética, esta subfamilia comprende cuatro géneros: Alphacoronavirus, Betacoronavirus, Gammacoronovirus y Deltacoronavirus de acuerdo a su estructura genética. Los alfacoronavirus y betacoronavirus infectan solo a mamíferos y normalmente son responsables de infecciones respiratorias en humanos y gastroenteritis en animales. Los coronavirus gamma y delta producen mayoritariamente infecciones en las aves y en los peces. Los coronavirus que afectan a los perros causan diarreas de gravedad variable, y los propios de los felinos pueden desencadenar la peritonitis infecciosa felina.
Estructuralmente los coronavirus son virus esféricos (fig. 1) de 80-160nm de diámetro, con envuelta de bicapa lipídica y que contienen ARN monocatenario de polaridad positiva de entre 26 y 32 kilobases de longitud y simetría helicoidal (11-13nm de diámetro) (1). El genoma de referencia para el SRAS-CoV-2 propuesto por GenBank posee 29.903 pares de bases (fig.2). Es el mayor entre los virus ARN, lo que explica su capacidad de recombinación genética con otros virus ARN.

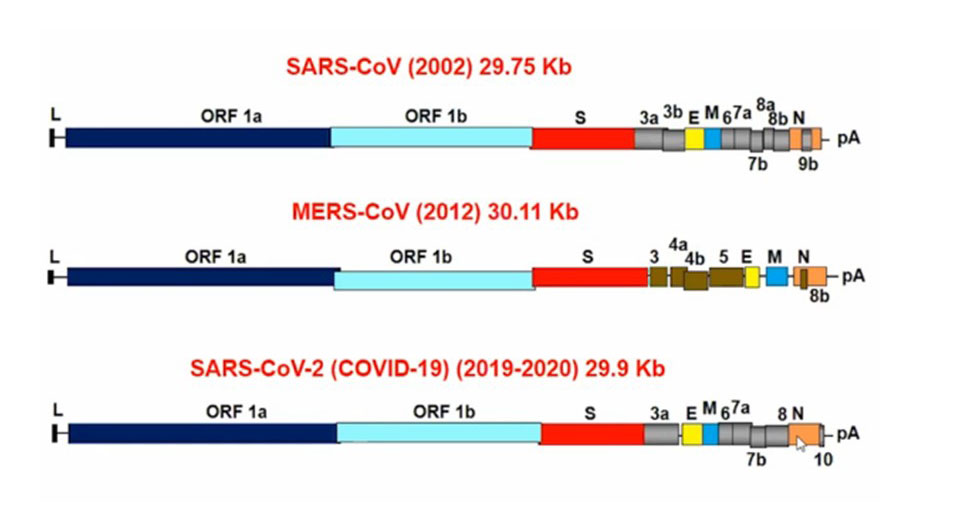
La organización del genoma tiene un orden característico: 5`-replicasa, gen de las espículas, gen de la envoltura, gen de la membrana, gen de la nucleocápside-3` (2).
Este genoma del virus SARS-CoV-2 codifica, por tanto, 4 proteínas estructurales: la proteína S (spike protein), la proteína E (envelope), la proteína M (membrane) y la proteína N (nucleocapsid).
La proteína N está en el interior del virión asociada al RNA viral, y las otras tres proteínas están asociadas a la envuelta viral. La proteína M de 25 Kdal está integrada en la membrana y es la proteína más abundante de la envoltura, y la proteína E, escasa, cuya función principal es el ensamblaje vírico. La proteína S forma estructuras de 20nm que sobresalen de la envuelta del virus, como pétalos, y conforman unas protrusiones muy espaciadas en la superficie de la partícula, que acaban en un copete más amplio, dando la forma de una corona, que es el origen del nombre de estos virus (figura 1). Esta glicoproteína S contiene dos subunidades, S1, que actúa como el ligando que se une a receptores de superficie de la célula diana, y S2, que interviene en la fusión de la envoltura del virión con la membrana citoplásmica de la célula (3).
Además, la proteína S sería la responsable como antígeno, de la producción de anticuerpos.
La glicoproteína S1 contiene el dominio de unión al receptor de las células que infecta, unión que se realiza por el dominio RBD (Receptor Binding Domain) y tiene lugar con el receptor celular ACE-2 (Angiotensin Converting Enzyme-2) (4-7).
Entre las células que poseen el receptor ACE-2 destacan las de la mucosa nasal y faríngea, la conjuntiva, los neumocitos pulmonares, las células intestinales y renales, el endocardio, testículos y células nerviosas cerebrales (8). Las dos primeras podrían ser la puerta de entrada del virus en los individuos.
El mecanismo de actuación del virus dentro de las células, aunque no está al 100% identificado molecularmente, sigue el patrón habitual de virus con envuelta (fig. 3):
- Adsorción. El virus, que se une a la célula hospedadora e introduce su material genético, es muy específico y únicamente infecta a un determinado tipo de células, las que poseen el receptor ACE-2, en el caso del SARS-CoV-2
- Penetración. La membrana de estos virus es de la misma naturaleza que la membrana celular, por lo que puede ocurrir una fusión de membranas, y entra sólo la cápside, o puede entrar por endocitosis, y la envuelta del virus se fusiona con el lisosoma.
- Decapsidación. El material genético queda libre en el citoplasma a través de diferentes enzimas que degradan las proteínas víricas.
- Síntesis y replicación. En el SARS-CoV-2, al ser un virus con ARN, esta fase ocurre solamente en el citoplasma. El virus utiliza la maquinaria celular para su replicación (creación de copias) del ARN y para la síntesis de las cuatro proteínas que ya hemos comentado.
- Ensamblamiento. En este momento, la célula posee copias de ARN del virus y de proteínas flotando en el citoplasma. Por diferentes mecanismos, y la actuación de la proteína E, estas proteínas se van uniendo dejando en su interior una copia del ARN viral. Las nuevas proteínas estructurales y accesorias recién sintetizadas son transferidas desde el retículo endoplasmático al aparato de Golgi, donde se ensamblan los nuevos viriones.
- Liberación. Tiene lugar por mecanismos de exocitosis, lo que les facilita rodearse de membrana. Es decir, la membrana del los virus con envuelta se origina de la membrana celular a la que ha infectado (9).
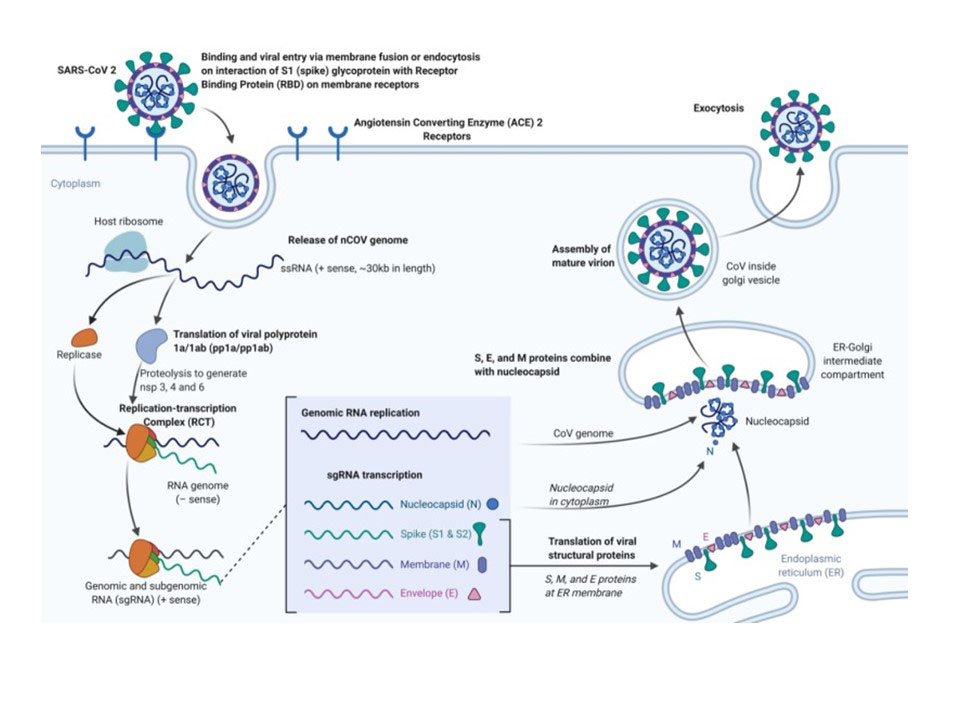
El virus, una vez en el interior de las células parasitadas podría desencadenar una reacción inmunológica (véase más adelante), pero también se ha comprobado que inhibe la producción y el mecanismo de acción del interferón, respuesta defensiva del organismo frente a todos los virus.
Los coronavirus son difíciles de cultivar a partir de muestras de los enfermos. Algunas cepas se han desarrollado en cultivos de células diploides humanas, pero la mayoría se han aislado en cultivos de células de tráquea humana embrionaria. Su crecimiento se demuestra por el cese del movimiento ciliar y la observación de los virus por microscopía electrónica o inmunomicroscopía electrónica. Sin embargo, algunas cepas se han podido adaptar en cultivos celulares, lo que ha permitido la práctica de encuestas serológicas en la población, mediante reacciones de neutralización, fijación de complemento y ELISA. Mediante la primera se pudo comprobar ya en 1980, que alrededor del 50% de los niños de 5-7 años y el 80% de los adultos presentaban anticuerpos, que indican infecciones anteriores (10).
Otro aspecto de gran interés es la relación genética de los coronavirus entre sí, para conocer el posible origen de las epidemias. Desde que en 1965, Tyrre y Bynoe cultivaron un virus obtenido de las vías respiratorias de un niño con resfriado común (2), gran número de trabajos comprobaron partículas similares en pollos, ratas, ratones, pavos y otras aves, bovinos, camellos y dromedarios, rumiantes salvajes, ballenas beluga, perros, gatos, conejos y cerdos, que daban manifestaciones clínicas muy variadas. Pero pronto los estudios comprobaron que la relación genética más cercana era con los virus de los murciélagos. Esta cercanía genética y su capacidad de recombinación se comprobaron en el SARS-CoV-1 con los virus de los murciélagos y civetas, y en el MERS-CoV con los de murciélagos y dromedarios (11). En el caso del virus SARS-CoV-2, la homología del ARN con el SARS-CoV-1 es del 60%, (fig. 2), pero con el BatCoV-RaTG13 hallado en poblaciones de los llamados murciélagos de herradura de la provincia de Yunnan (China) (Rhinolophus affinis) es del 96,2%. Sin embargo, el hecho de que este virus del murciélago presente diferencias en más de 1.100 nucleótidos con el SARS-CoV-2 plantea dudas sobre que pueda ser su origen inmediato, y sugiere la necesidad de que haya evolucionado en otro hospedador intermediario. El virus del pangolín malayo posee una similitud el 91.02% con el genoma del SARS-CoV-2 y por ello, para algunos autores, ese podría ser el huésped intermediario (12).
Técnicas de laboratorio
Diagnóstico directo para la búsqueda del virus
La técnica de referencia es la RT-PCR o reacción en cadena de la polimerasa, en la que se detecta el ARN del virus, y permitiéndonos la cuantificación mediante la determinación de la carga vírica (13). Normalmente se hace mediante toma con hisopo de secreciones nasofaríngeas. Recientemente, en un estudio realizado en individuos que acudieron a un hospital por una infección respiratoria aguda se realizaron dos tomas, una con hisopo nasal y otra en saliva. La prevalencia hallada en la muestra respiratoria fue del 9,5% y la especificidad fue del 84.2%. La concordancia entre ambas tomas fue del 97.5%. El estudio en saliva, podría ser una muestra alternativa y, según sus autores, facilitaría la recolección de muestras y la mejoría en el diagnóstico (14).
Detección del antígeno
La detección del llamado antígeno vírico, que también se realiza con un hisopo a partir de nasofaringe, consiste en el hallazgo de cualquier fragmento de las proteínas de la superficie del virus, mediante inmunoensayo fluorescente (SOFIA-2). Ha sido estudiado en la corporación Quidel de San Diego, y aprobado por la FDA (15). Se necesita captar una gran cantidad de proteínas para poder detectar una infección positiva, lo que explica que existan un mayor número de falsos negativos. Una de las ventajas es la rapidez de la técnica, que se puede llevar a cabo en 15 minutos.
Detección de anticuerpos
Detección rápida: TDR. Consiste en una inmunocromatografía, similar a la prueba de embarazo, ya que muestra líneas de color para los sueros positivos. Permite detectar IgM e IgG (fig. 4). Son sencillas y rápidas. Pero con una sensibilidad que permite el escape de casos positivos, lo que explicaría el elevado número encontrado de casos negativos. En estudios comparativos de los test rápidos y el ELISA se demostró la alta variabilidad de los primeros (13).
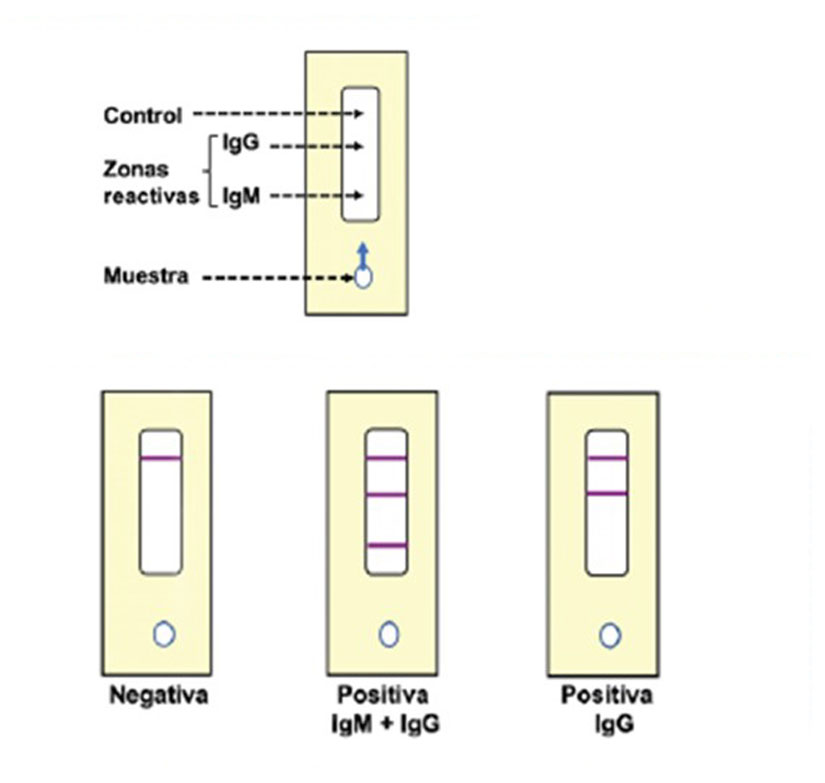
La técnica de referencia es el ELISA (enzimoinmunoanálisis). Además de detectar IgM e IgG, permite la cuantificación de los anticuerpos. Utiliza una placa de pocillos recubiertos por la proteína S del virus, que se incuban con el suero a estudiar. Es muy sensible y específica.
Cinética de las pruebas diagnósticas
En la figura 5 aparece reflejada la cinética de la PCR y de los anticuerpos en la infección por el virus. Tras el contacto con el virus comienza un período asintomático que puede durar varios días, en el cual raramente se utiliza ninguna técnica diagnóstica. Con el inicio de los síntomas podemos detectar el ARN por PCR, que inicia su aumento, descendiendo posteriormente a medida que el sujeto comienza a recuperarse, desapareciendo a los 20-21 días. Esta prueba de diagnóstico busca fragmentos genéticos de una infección activa o menguante. Un resultado positivo después de la desaparición de los síntomas, no indica necesariamente una reinfección. Se han descrito PCR positivas pasados 28 días.

Sobre los días 6 o 7 comienza la respuesta de anticuerpos, siendo la IgM la primera. A los 14-17 días aparece la IgG, que se eleva notablemente, manteniéndose en forma de meseta unos 35 días, aproximadamente. En pacientes hospitalizados y recuperados, las tasas de IgG son similares en hombres y mujeres, pero presentan un título más alto en relación a la gravedad del paciente, así como en ancianos, probablemente porque presenten una mayor gravedad (16). Estos anticuerpos pueden proporcionar una inmunidad a largo plazo.
Incertidumbres
Debido al elevado número de pruebas diagnósticas y a sus resultados poco concordantes, sobre todo, si lo relacionamos con los cuadros clínicos, surgen una serie de dudas y problemas en la interpretación de procesos, creando una verdadera incertidumbre. Dichas dudas son numerosas, pero las sintetizaremos en diversos apartados: Duración de la inmunidad, reinfecciones, capacidad real de la neutralización de los anticuerpos y aparición de nuevas patologías.
Duración de la inmunidad: Realmente no sabemos con certeza la duración de los anticuerpos y si la inmunidad que proporcionan los mismos es duradera. Tenemos como ejemplo la pandemia del SARS por el virus CoV-1, que desapareció de forma rápida, y de la cual se conservan sueros de pacientes que la padecieron y que nos puede hacer pensar en una inmunidad adecuada tras varios años. En el caso del SARS-CoV-2 el tiempo transcurrido es todavía demasiado corto para asegurar cualquier hipótesis.
Sobreinfección o reinfección: Puede deberse al contacto con otros coronavirus humanos, al fallo de una hipotética inmunidad protectora, a la exposición a formas genéticamente distintas de la misma cepa viral o al desprendimiento de fragmentos de ARN capaces de ser captados por la técnica de ensayo, dando lugar a resultados falsos positivos de la PCR (17).
Capacidad real de la neutralización de los anticuerpos:Para conocer esta capacidad con exactitud se requieren ensayos de neutralización, con suero o plasma de pacientes, y así comprobar enfrentándolos a virus, la inhibición de su capacidad replicativa.
Posible aparición de nuevas patologías: Se han denominado ADE (Antibody Dependent Enhancement) a procesos debidos a la fijación de la fracción Fc de anticuerpos no neutralizantes, capaces de estimular diversas células del sistema inmune. Serían un paso clave en la progresión de la enfermedad de su forma leve a severa (18). Así mismo, podrían explicar la disregulación inmune, incluida la apoptosis de células inmunes, que promueve el desarrollo de linfopenia de células T, y una cascada inflamatoria con la acumulación pulmonar de macrófagos y neutrófilos, así como, una tormenta de citocinas (19). Quizá el fenómeno ADE sea la razón por la cual el curso del SARS-CoV-2 es más severo en las personas mayores, en comparación con las más jóvenes, debido a que la producción de anticuerpos es en ellas más lenta y no suficiente para neutralizar los virus.
Otras posibles patologías que puedan darse, como fibrosis pulmonar, daños intestinales, renales, cerebrales, dermatológicos, etc. deben de ser considerados en un futuro próximo.
¿Qué es lo que podemos y debemos buscar para solucionar los problemas planteados?
Algunos de los grados de incertidumbre encontrados desde el comienzo de la pandemia de SARS-CoV-2 hasta ahora, han ido creciendo conforme pasaba el tiempo y, a pesar de existir numerosos estudios de todo tipo en todo el mundo y por ello, una amplia bibliografía, es absolutamente necesario que transcurra aún más tiempo para encontrar soluciones adecuadas.
La formación de grupos de investigación formados por microbiólogos, inmunólogos, clínicos y epidemiólogos, con un seguimiento conjunto en amplias cohortes de todos los resultados, es la única solución a los problemas que hemos planteado, así como todos aquellos que de hecho puedan aparecer y así podremos profundizar en las innumerables incógnitas científicas.
BIBLIOGRAFÍA
- Chen Y, Liu Q, Guo D. Emerging coronaviruses: genome structure, replication, and pathogenesis. J Med Virol 2020; 92 (4): 418-423.
- Maroto MC, Piédrola G. Los coronavirus. An RANM 2019; 136(03): 235-238.
- Nal B, Chan C, Siu L. Differential maturation and subcellular localization of severe acute respiratory syndrome coronavirus surface proteins S, M and E. J Gen Virol 2005; 86: 1423-1434.
- Kuhn JH, Li W, Choe H, Farzan M. Angiotensin-converting enzyme 2: a functional receptor for SARS coronavirus”. Cell Mol Life Sci 2004; 61(21): 2738-2743.
- Zhang H, Penninger JM, Li Y, Zhong N, Slutsky A. Enzima convertidora de angiotensina 2 (ACE2) como receptor de SARS-CoV-2: mecanismos moleculares y posible objetivo terapéutico. Cuidados Intensivos Med 2020; 46 :586-590.
- Kai H, Kai M. Interactions of coronaviruses with ACE2, angiotensin II, and RAS inhibitors : lessons from available evidence and insights into COVID-19. Hypertens Res 2020; 43: 648-654. https://doi.org/10.1038/s41440-020-0455-8
- Sama IE, Ravera A, Santema BT et al. Circulating plasma concentrations of angiotensin-converting enzyme 2 in men and women with heart failure and effects on renin-angiotensin-aldosterone inhibitors. Eur Heart J 2020; 41 (19): 1810-1817. doi:10.1093/eurheart/ehaa.373.
- Bunyavanich S, Do A, Vicencio A. Nasal gene expression of angiotensin-converting enzyme 2 in children and adults. JAMA 2020. doi: 10.1001/jama.2020.8707.
- Cascella M, Rajnik M, Cuomo A, Dulebohn SC, Di Napoli R. Features, evaluation and treatment coronavirus (COVID-19). StatPearls. https://www.ncbi.nlm.nih.gov/books/NBK554776/
- Pumarola A. Coronavirus. En: Pumarola A, Rodríguez-Torres A, García-Rodríguez JA, Piédrola G. Microbiología y parasitología médica. 2ª ed. Barcelona: Salvat, 1987, p. 691-692.
- Coleman CM, Friemann MB. Coronaviruses: important emerging human pathogens. J Virol 2014; 88(10): 5209-5212.
- Zhang T, Wu Q, Zhang Z. Probable pangolin origin of SARS-CoV-2 associated with the COVID-19 Outbreak. Curr Biol 2020; 30(7): 1136-1341.
- Martínez L, García F, Navarro JM, Pascual A. Recomendaciones de la Sociedad Andaluza de Microbiología y Parasitología Clínica para el uso de los test de detección de antígeno o de detección de anticuerpos para el diagnóstico de la infección por SARS-CoV-2. SAMPAC, 4 de Abril 2020.
- Pasomsub E, Siriorn P, Boonyawat K et al. Saliva sample as a non-invasive specimen for the diagnosis of coronavirus disease 2019: a cross-sectional study. Clin Microbiol Infect. 2020 https://doi.org/10.1016/j.cmi.2020.05.001
- Jacobs A. F.D.A. approves first antigen test for detecting the Coronavirus. The New York Times, May 11, 2020.
- Stadlbauer D, Amanat F, Chromikova V et al. SARS-CoV-2 seroconversion in humans: a detailed protocol for a serological assay, antigen production, and test setup. Curr Protoc Microbiol 2020; 57. e100. doi: 10.1002/cpmc.100
- Hyun-ju O. 91 recovered COVID-19 patients test positive again: KCDC. The Korea Herald. 2020, 10 Abril.
- Yip MS, Cheung CY, Li PH et al. Investigación de mejora dependiente de anticuerpos (ADE) de la infección por coronavirus SARS y su papel en la patogénesis del SARS. Actas de BMC 2011; 5 (S1). doi : 10.1186 / 1753-6561-5-s1-p80 .
- Wan Y, Shan J, Sun S. et al. Mecanismo molecular para la mejora dependiente de anticuerpos de la entrada de coronavirus. J Virol 2020; 94(5): e02015-19.
DECLARACIÓN DE TRANSPARENCIA
El autor/a de este artículo declara no tener ningún tipo de conflicto de intereses respecto a lo expuesto en el presente trabajo.
ranm tv
María del Carmen Maroto Vela
Real Academia Nacional de Medicina de España
C/ Arrieta, 12 · 28013 Madrid
Tlf.: +34 91 159 47 34 | Email de correspondencia
Año 2020 · número 137 (02) · páginas 98 a 103
Enviado*: 26.05.20
Revisado: 30.05.20
Aceptado: 14.06.20
* Fecha de lectura en la RANM


