Abstract
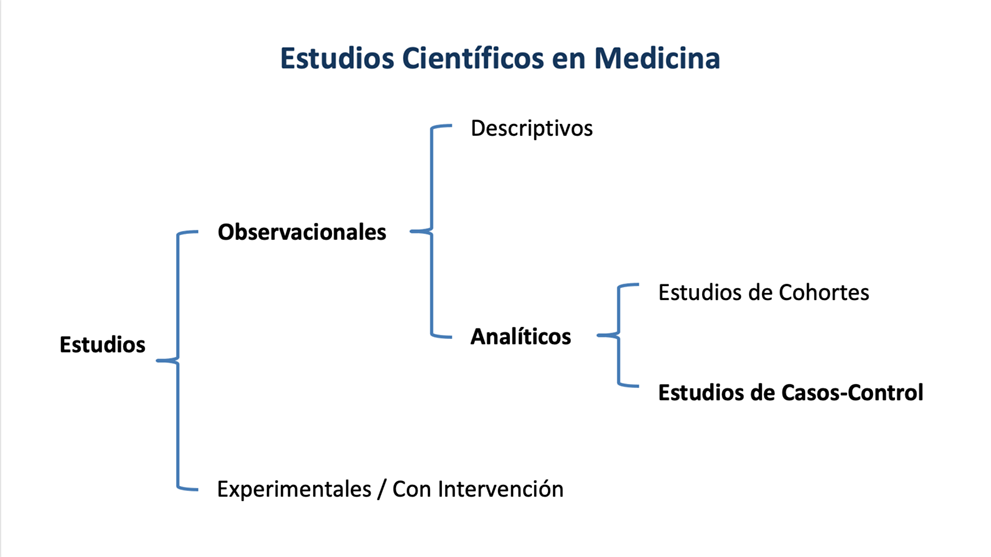
Este trabajo define una estrategia de investigación con datos (research on data) centrada en la imagen médica y sus biomarcadores de imagen derivados para finalmente evaluar de forma crítica el concepto de inferencia causal y valorar sus incertidumbres. En el desarrollo de este trabajo se pondrán en valor los estudios observacionales computacionales para generar inferencias de causalidad en el mundo real (real world data, real world evidence). La finalidad última es proponer una metodología científica que permita estimar causalidades con estudios observacionales mediante el control de calidad de las bases de datos, la definición de hipótesis plausibles, utilizando modelos computacionales estimativos e inteligencia artificial. La aproximación computacional de la radiología a la medicina de precisión utiliza estrategias epidemiológicas con estudios de inferencia causal, utilizando datos del mundo real con una metodología observacional, longitudinal y de análisis con casos y controles (siendo caso la presencia, y control la ausencia del evento que se quiere estimar). En este entorno, la consideración de enfermedad en epidemiología clásica se traslada ahora a fenotipado, respuesta al tratamiento y pronóstico final; y la exposición a un factor se considera como presencia de un biomarcador de imagen radiómica, dinámico o un modelado con inteligencia artificial. La investigación con datos para inferir causalidad se realiza sobre un conjunto de casos cerrados reclutados, en los que el investigador no interviene en la historia clínica del paciente pero trabaja sobre datos para su uso secundario en la generación de causalidades consistentes.
Resumen
This work defines a research on data strategy focused on medical imaging and derived image biomarkers to critically assess the concept of causal inference and uncertainties. Computational observational studies will be valued to generate casual inference from real world data. Our main goal is to propose a scientific methodology that allows to estimate causalities from observational studies through quality control of large databases, definition of plausible hypotheses, using computational estimated models and artificial intelligence tools. The computational approach of radiology to precision medicine by using epidemiological strategies is based on causal inference studies relies on real-world data observational, longitudinal, case-control analysis designed (being case the presence, and control the absence of the event to be estimated). In this new research setting, we consider disease in classical epidemiology as phenotyping, response to treatment and final prognosis; and exposure equals to the presence of a radiomic, dynamic image biomarker or AI modeling solution. Research with data on which causality is to be inferred must control for recruitment of closed cases, in which the researcher does not intervene in the patient’s clinical history but works on databases, collecting data to be secondary used in generating consistent causalities.
Keywords: Estudios Observacionales; Inferencia Causal; Biomarcadores de Imagen; Radiómica; Datos del Mundo Real.
Palabras clave: Observational Studies; Causal Inference; Imaging Biomarkers; Radiomics; Real World Data.
INTRODUCCIÓN
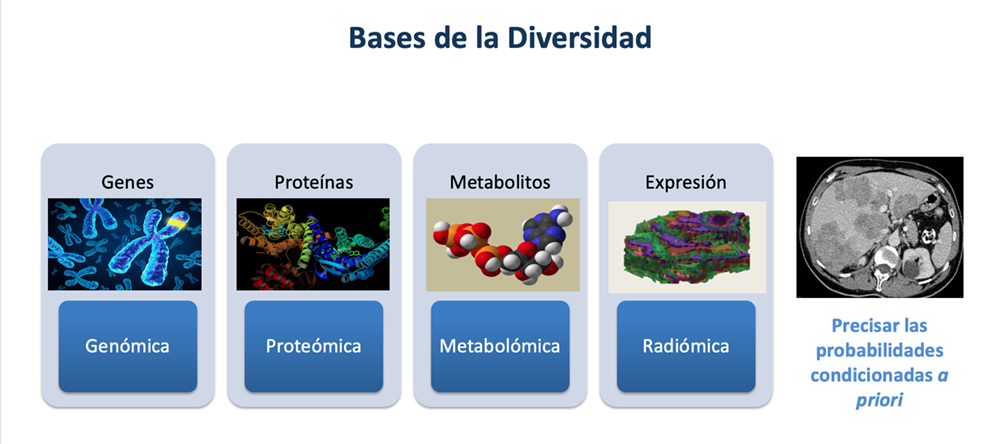
La investigación es inherente a la medicina, ya que se requiere evidencia para saber cómo se comportará una enfermedad y cuál será el resultado de un tratamiento. Nuestra diversidad biológica es la base principal de la heterogeneidad en el comportamiento de las lesiones y la evolución de las enfermedades que nos aquejan, justificando esta variabilidad los diferentes comportamientos entre pacientes con una misma enfermedad. Nuestra diversidad funcional se explica tanto por las diferencias genéticas como por las de expresión fenotípica tanto entre las lesiones como en los pacientes (Figura 1). Es por ello por lo que la Medicina de Precisión analiza esta variabilidad para predecir con una mayor certeza el comportamiento esperado de una lesión en un paciente determinado.
Una excelente aproximación para captar esta interacción entre la situación particular de un paciente y la expresión de su enfermedad es la evidencia generada mediante los estudios observacionales analíticos tipo caso-control. Estas investigaciones se diseñan como estudios realizados no con pacientes sino con sus datos obtenidos de entornos clínicos habituales. Utilizan para ello repositorios bien estructurados que incluyen tanto las imágenes como los datos clínicos y moleculares, para generar modelos analíticos con información consistente y coherente (1). Se reconoce en esta aproximación que diseñar estudios científicos observacionales para definir relaciones de causa-efecto en la Medicina de Precisión requiere de una gran cantidad de información cualificada, obtenida del mundo real, sobre la que implementar herramientas computacionales (radiómica y biomarcadores de imagen) y soluciones de Inteligencia Artificial. Se pretende de esta forma que las conclusiones extraídas de los estudios sean reproducibles y estén validadas, minimizando el problema del submuestreo, la heterogeneidad de la muestra analizada y la variabilidad por falta de estandarización de los datos y las imágenes analizadas.
Nuestro entorno científico pretende aproximarse a la Medicina de Precisión desde la imagen computacional a través de la radiómica y los biomarcadores de imagen. Muy diversas aproximaciones previas se han definido previamente para entornos y situaciones clínicas muy diferentes (2-10). En general, para evaluar la situación clínica de muchos pacientes se obtienen imágenes digitales mediante diferentes modalidades (tales como radiografía, ecografía, tomografía computarizada, resonancia magnética y tomografía por emisión de positrones). Estas imágenes pueden procesarse con algoritmos computacionales y redes neuronales para extraer diversos parámetros no observables directamente, y que pueden tener una relación de causalidad para predecir la probabilidad de que suceda un evento clínico final que consideramos relevante a la enfermedad.
La radiómica permite la extracción computacional de datos cuantitativos a partir de estas imágenes adquiridas en la vida real para conocer la expresión de diferentes propiedades tisulares previamente relacionadas mediante modelos computacionales o redes neuronales convolucionales (Deep features). Estos datos cuantitativos provienen del análisis de los diferentes atributos de la propia imagen o de la modificación dinámica de la señal al cambiar algún parámetro en la adquisición (por ejemplo, en los estudios de difusión en RM o los análisis espectrales en TC). Se considera que los parámetros radiómicos obtenidos son biomarcadores de imagen cuando estén subrogados a una propiedad de la lesión o un devenir clínico del paciente. Con estos datos radiómicos pueden construirse modelos descriptivos y predictivos de una realidad biopatológica o una situación clínica de interés (clinical endpoint). Si la relación cumple criterios de causalidad habremos establecido una inferencia causal (11).
Para inferir causalidad debemos consensuar los términos relacionados que se utilizarán en este trabajo. Así, puede considerarse como causa toda condición o característica que aumenta la probabilidad de ocurrencia de un evento, como puede ser el desarrollo de una enfermedad o la respuesta a un tratamiento, mientras otras condiciones de exposición permanecen fijas. Un efecto causal será el desenlace observado en un individuo o una población tras una intervención o una exposición, y que necesariamente es diferente del que se observaría en ausencia de esa intervención o exposición. En este contexto, se considera como intervención a cualquier acción realizada a un paciente que no se habría realizado fuera del estudio de causalidad, como puede ser un diagnóstico o tratamiento fuera de práctica habitual. Por otro lado, se considera exposición a cualquier factor o característica de un paciente que se entiende pueda ser un factor causal, como por ejemplo una expresión radiómica determinada en una lesión tumoral.
Con estas premisas, entendemos la inferencia causal como el proceso establecido para determinar si es causal la relación entre una intervención o exposición y un evento clínico de interés como la predicción de respuesta a un tratamiento. Esta causalidad puede ser el resultado de relacionar varias causas para ser responsable suficiente del evento observado con posterioridad. Es muy oportuno reconocer aquí la complejidad de las relaciones de causalidad, dado que una causa puede ser necesaria, pero también solo facilitadora, y puede ser primaria, pero también derivada o intermedia. Además, y como hemos mencionado, con frecuencia se observa un fenómeno de multicausalidad por el que se reconoce que el efecto final responde a diversas causas explicativas relacionadas entre sí.
Reconocer la multicausalidad es apreciar los límites para atribuir causalidades únicas con certeza y desarrollar el concepto de firmas nosológicas tisulares.
Además, para atribuir causalidad con un nivel alto de certeza y evidencia debe reconocerse que los modelos sobre los que se sostiene pueden estar sobreajustados, es decir, ser excelentes sólo para el conjunto de datos con los que se han creado y entrenado mientras que fallarán cuando se extrapolen a poblaciones diferentes. En la atribución de causalidad también debemos considerar el principio de parsimonia, conocido igualmente como la navaja de Ockham, según el cual la pluralidad no debe postularse si no es necesaria; es decir, las explicaciones deben ser lo más simples posibles mientras se mantenga la potencia causal.
En este sentido, es relevante reconocer que empíricamente se acepta que la dimensionalidad de los datos explicativos en medicina no suele ser mayor de cinco (valor atribuido al estadístico John Tukey), limitándose así la dimensión de la multicausalidad.
Diversidad y Causalidad
Como base de la Medicina de Precisión se reconoce en la actualidad que las personas y sus enfermedades son modelos dinámicos con diferencias significativas que se expresan como variables bayesianas, es decir, representando la disposición o probabilidad previa a desarrollar un fenómeno biopatológico o un evento clínico determinado. Estas expresiones son en el fondo indicadores probabilísticos de los resultados finales que se quieren estimar. De alguna forma, los modelos causales simulan la arquitectura de una red neuronal, donde las variables iniciales (inputs) interactúan causalmente para generar unos resultados finales (outputs) que son diferentes para cada individuo dadas las diferencias en las variables iniciales. Desvelar estos Biomarcadores Multiescala iniciales, es decir, estas exposiciones que interactúan para generar una respuesta, permitirá establecer mejores modelos predictores del comportamiento de una enfermedad. Reconociendo esta complejidad biológica se pretende establecer inferencias de causalidad para predecir la agresividad, la respuesta y el pronóstico más probables en un paciente dado con una expresión inicial a priori conocida. Estas expresiones iniciales de partida son los Biomarcadores Multiescala que pretendemos relacionar con las respuestas clínicas de interés a través de los modelos estimativos. El concepto multiescala se relaciona con las diferentes expresiones principales (genómica, proteómica, metabolómica, radiómica) que constituyen el elenco de posibles inferencias causales que son la base de la biodiversidad reconocida por la Medicina de Precisión (Figura 1).
Como ejemplo de relación de causalidad entre la imagen computacional y la estimación de eventos futuros podemos introducir ahora la Radiogenómica. Las diferencias genotípicas, los distintos perfiles genéticos y las posibles mutaciones originan diferencias de expresiones y comportamientos. Así, el color del iris tiene un determinante genético. Las características y propiedades internas de algunas alteraciones y lesiones pueden también considerarse como expresiones fenotípicas externas en los tumores y lesiones. Así, los diferentes hábitats que se observan en muchos tumores heterogéneos pueden identificarse mediante la agrupación de datos radiómicos y los modelados dinámicos analizados mediante imagen computacional. Estos parámetros de la apariencia de las lesiones derivados del análisis de imágenes serán biomarcadores de imagen (univariantes) o firmas nosológicas tisulares (multivariantes) cuando se relacionen con modificaciones concretas en su expresión biológica, en su respuesta a un tratamiento dado o en su evolución clínica. Así, se ha observado que ciertas características de la textura de un tumor, caracterizada por la variación espacial de sus niveles de gris en la lesión y captados a través de parámetros como su Entropía, Inercia, Energía, Correlación, Homogeneidad, Uniformidad y Fractales, se relacionan con la supervivencia general del paciente en algunos tipos de cáncer como el de endometrio, próstata y glioblastoma (2,8,9,12,13,14). Es decir, la apariencia de una lesión es un predictor de la respuesta al tratamiento y de la supervivencia general al momento del diagnóstico inicial. Es por ello evidente que estas variables radiómicas deberían considerarse para clasificar por probabilidad bayesiana las cohortes longitudinales de pacientes a las que se pretende administrar un fármaco cuya eficiencia queremos valorar. Sólo de esta forma puede evitarse el sesgo de probabilidad diferencial que interferiría en la estimación de inferencia causal (Figura 2).

Como hemos mencionado, las inferencias causales implican con frecuencia la imbricación de diferentes variables iniciales explicativas. En los desarrollos con imagen computacionales se conoce como Firma Nosológica a la estimación multiparamétrica que incluye diversas métricas radiómicas para agruparlas en un único parámetro que se representa como la distribución espacial resuelta en heterogeneidad de una inferencia causal con un evento final. Para cada evento biopatológico o pronóstico se busca una firma nosológica extraída de las imágenes que prediga su ocurrencia (12-14).
Epidemiología Experimental y Medicina de Precisión
Los avances en medicina se han realizado generalmente a través de los estudios experimentales prospectivos. Estos Ensayos Clínicos intervienen modificando el devenir de la enfermedad y suelen construirse comparando si se produce un efecto cuando se realiza una intervención (un tratamiento, una exposición) frente a un grupo control en el que no se realiza esa intervención (aunque se pueda simular con un placebo). Los pacientes suelen distribuirse de forma aleatoria entre los dos grupos y los pacientes en los grupos no presentan diferencias en factores generales (como la edad o el género) que pudieran ser variables de confusión. En este sentido, el paradigma de estudio con mayor validez es el ensayo clínico controlado y aleatorizado en el que los sujetos estudiados representan la población diana sobre la que se quiere conocer el efecto de causalidad de una intervención dada. Estos ensayos clínicos se construyen con un control muy alto de los datos que se adquieren, su homogeneidad y estandarización interna están verificadas, y su aceptación científica y nivel de evidencia es muy alto. Como desventaja, podemos señalar que la población incluida en estos ensayos presenta un tamaño muestral limitado, la duración del estudio está acotada en el tiempo, sus criterios de inclusión son muy restrictivos con exclusión de comorbilidades, y tiene un sesgo alto de submuestreo de la población estudiada y presentan un alto coste económico y en recursos de personal involucrado (15).
Por contra, un estudio observacional sin intervención sobre datos del mundo real tiene la ventaja de construirse con un mayor muestreo poblacional, incluyendo grupos de población mayores y covariables de comorbilidad, con un menor coste económico y de investigadores involucrados (Figura 3). Con estos estudios se pretende emular los ensayos de investigación incluyendo pacientes del mundo real y aprovechando la información disponible en las historias clínicas digitales (16). Aunque estos estudios tienen una menor aceptación científica, para extraer inferencias causales válidas debe controlarse adecuadamente la mayor heterogeneidad y minimizar la falta de estandarización en la calidad de los datos en origen. Por ejemplo, debemos normalizar las imágenes médicas dada su alta variabilidad en los protocolos de adquisición.
Emular Estudios Experimentales con Datos del Mundo Real para Inferir Causalidad
Para establecer razonablemente las relaciones de causalidad en los estudios observaciones deben tenerse en cuenta una serie de requisitos para emular la construcción de un estudio prospectivo (17). Así, deben definirse las variables que se van a recoger en la base de datos del estudio, con su descripción y relevancia. Debe analizarse la distribución y representatividad de la población diana que se quiere estudiar. Por otro lado, debe considerarse la minimización del número de variables a aquellas relevantes y eliminar las variables redundantes para evitar las asociaciones causales espurias.
Los datos recogidos deben estratificarse como variables descriptoras de la población diana, variables predictoras (o de entrada) y variables de respuesta (o de salida) del modelo. Las variables recogidas deben constituirse en un Conjuntos de Datos Básicos Mínimos para los objetivos que se hayan definido en el estudio, siguiendo criterios de evidencia científica. Deben emplearse los estándares que faciliten la extracción universal de datos de las historias clínicas (OMOP-CDM, Common Data Model), identificando y eliminando los elementos redundantes (18).
Para garantizar una universalidad y reproducibilidad aceptable de los resultados debe calcularse inicialmente el tamaño muestral mínimo para evitar el sesgo del submuestreo. Debe también ajustarse la potencia estadística necesaria aumentando la fuerza de asociación con criterios más exigentes (tales como un p-valor<0.005 y el uso de las correcciones por FDR, False Discovery Rate) que eviten los sesgos de serendipia. Debe analizarse además la magnitud del efecto inducido, su relevancia y la coherencia con lo ya conocido, además de validar el resultado en otras series de casos tanto similares (validación interna) como diferentes (validación externa). De esta forma se emularán los diseños experimentales acotando el problema de la investigación con datos mediante un control de la selección y la evaluación de las variables (19, 20, 21).
Estos diseños observacionales analíticos con casos y controles deben seguir todavía más reglas para su aceptación como inferencia causal (1, 22, 23). Así, para poblar la base de datos que hemos definido con ese conjunto mínimos de datos relevantes deben seleccionarse los pacientes de una población diana con unos criterios de inclusión definidos durante el diseño del estudio. En concreto, debe establecerse si el reclutamiento está basado en la presencia de síntomas, en el resultado de pruebas de referencia, en la probabilidad de padecer una enfermedad o en la existencia de una enfermedad con unas características clínicas concretas basadas en estándares de referencia. Este reclutamiento de datos debe definirse si se realizará de forma consecutiva, en un centro o en varios, durante que periodo de tiempo y desde que entorno clínico de selección (primaria, especializada, referencia). También hay que definir en este momento los criterios de exclusión que se aplicarán para obtener una población final adecuada al estudio.
Es muy importante el control de las variables de confusión. Durante el diseño de la base de datos hay que definir las características sistemáticas que puedan influir en el resultado final y que se incluirán en la base de datos, tales como las diferencias epidemiológicas relevantes a la enfermedad, los diferentes fenotipos conocidos, los grados de expresión y los estadios de extensión de la enfermedad, los instantes temporales en los que se van a recoger los datos de cada paciente y los tratamientos que se recogen, incluyendo aquellos concomitantes. También es fundamental definir y justificar los patrones de referencia que van a emplearse, su descripción y su aceptación científica.
Para las variables más estructuradas relacionadas con los marcadores moleculares, genéticos y de imagen es fundamental definir de dónde, cómo y cuándo se obtuvieron los datos y sus instantes temporales. Debemos definir los biomarcadores que se extraerán de las imágenes como datos radiómicos. En definitiva, debe poder defenderse que la recogida de estos datos permitirá emular la recogida de datos de un proyecto o EECC prospectivo estándar. En este proceso debe tenerse un especial cuidado en la definición de las unidades, los puntos de corte y las categorías o grados de los clasificadores empleados, así como en la descripción de todos los tratamientos recibidos. También debe definirse el número, la capacitación y la experiencia de los observadores que manejan los datos, obtienen los biomarcadores e interpretan las variables poco estructuradas como las que sólo se recogen como texto libre en las historias clínicas. También debe hacerse constar si los lectores de las pruebas y del patrón de referencia conocían los resultados de las otras pruebas o los resultados clínicos finales a estimar; es decir, si el estudio es ciego.
En las inferencias de relación causa-efecto deben considerarse algunos de los criterios de causalidad de Bradford-Hill. De entre los más relevantes cabe resaltar que la relación inferida debe ser coherente (fiel a la biología y fisiopatología conocidas), potente y con gradiente (con magnitud y la dirección proporcionales), temporal (la causa precede al efecto y su curso evolutivo) y consistente (reproducible por diferentes investigadores y en diferentes entornos).
Finalmente, deben seguirse las recomendaciones estructurales estandarizadas para las publicaciones con modelos diagnósticos, predictores y pronósticos. Para que los resultados sean reproducibles y generalizables se recomienda el uso de los estándares STARD (eficacia diagnóstica), STROBE (estudios observacionales) y TRIPOD (modelos predictivos multivariantes) (24).
Esta metodología es la que se está siguiendo en el proyecto europeo Chaimeleon (Horizon 2020 Research and Innovation Programme No. 952172) del que somos coordinadores. En este proyecto se está creando un repositorio pan-Europeo de imágenes médicas y datos clínicos asociados de pacientes con cáncer de mama, próstata, pulmón y colorrectal como recurso abierto y en la nube. Este repositorio de datos debe servir para desarrollar herramientas de inteligencia artificial enfocadas a asistir en una mejor precisión del diagnóstico, pronóstico y tratamiento de estos pacientes (Figura 4). Estas herramientas de inteligencia artificial afectarán tanto al tratamiento de datos (como la segmentación automática de lesiones o la armonización de las imágenes para normalizar en un marco de trabajo homogéneo) como a la inferencia de modelos explicativos de causalidad (por qué en un mismo tipo de tumor unas lesiones se comportan de forma diferente a otras) (25).
CONCLUSIONES
El desarrollo de la salud digital, las historias clínicas electrónicas, los sistemas de almacenamiento masivo de imágenes y los biomarcadores de imagen han permitido a la radiología profundizar en el análisis de datos y la generación de inferencias causales. Así, la imagen computacional y la inteligencia artificial generan modelos predictores que integran datos clínicos, moleculares y de imagen. Estos modelos deben impactar en la asistencia a través de la evidencia obtenida del mundo real, entendida como el análisis de los datos generados en la práctica clínica habitual y su estudio con un diseño observacional longitudinal casos-control diseñado con un alto nivel de pragmatismo.
Estas investigaciones sobre datos, observacionales y longitudinales, permiten demostrar la existencia de relaciones y asociaciones entre una acción y un resultado, pero también avanzar en las inferencias de causalidad con un alto nivel de evidencia científica (26).
BIBLIOGRAFÍA
- Martí-Bonmatí L. La radiómica y los biomarcadores de imagen en los estudios clínicos observacionales con datos retrospectivos. Anales RANM 2019;136 (01):34-42.
- Rodríguez-Ortega A, Alegre A, Lago V et al. Machine learning-based integration of prognostic magnetic resonance imaging biomarkers for myometrial invasion stratification in endometrial cancer. J Magn Reson Imaging. 2021 (doi: 10.1002/jmri.27625. Epub ahead of print).
- Cerdá Alberich L, Sangüesa Nebot C, Alberich-Bayarri A et al. A confidence habitats methodology in MR quantitative diffusion for the classification of neuroblastic tumors. cancers (Basel). 2020;21;12(12):3858.
- Ballester-Vallés C, Flores-Méndez J, Delgado-Moraleda J et al. Hepatic and pancreatic fat as imaging biomarkers of metabolic syndrome. Radiologia. 2020;62(2):122-130.
- Mazón M, Vázquez Costa JF, Ten-Esteve A, Martí-Bonmatí L. Imaging biomarkers for the diagnosis and prognosis of neurodegenerative diseases. The example of amyotrophic lateral sclerosis. Front Neurosci. 2018;25(12):784.
- Vázquez-Costa JF, Mazón M, Carreres-Polo J et al. Brain signal intensity changes as biomarkers in amyotrophic lateral sclerosis. Acta Neurol Scand. 2018;137(2):262-271.
- Alberich-Bayarri Á, Hernández-Navarro R, Ruiz-Martínez E et al. Development of imaging biomarkers and generation of big data. Radiol Med. 2017;122(6):444-448.
- Sanz-Requena R, Martí-Bonmatí L, Pérez-Martínez R, García-Martí G. Dynamic contrast-enhanced case-control analysis in 3T MRI of prostate cancer can help to characterize tumor aggressiveness. Eur J Radiol. 2016;85(11):2119-2126.
- Sanz-Requena R, Revert-Ventura A, Martí-Bonmatí L, Alberich-Bayarri A, García-Martí G. Quantitative MR perfusion parameters related to survival time in high-grade gliomas. Eur Radiol. 2013;23(12):3456-3465.
- Martí Bonmatí L, Alberich-Bayarri A, García-Martí G et al. Biomarcadores de imagen, imagen cuantitativa y bioingeniería. Radiología. 2012; 54(3):269-278.
- Rothman KJ, Greenland S. Causation and causal inference in epidemiology. Am J Public Health. 2005;95(Suppl 1):S144-50.
- Juan-Albarracín J, Fuster-Garcia E, Pérez-Girbés A et al. Glioblastoma: vascular habitats detected at preoperative DSW CE-perfusion MR imaging predict survival. Radiology 2018;287(3):944-954.
- Fuster-Garcia E, Juan-Albarracín J, García-Ferrando GA, Martí-Bonmatí L, Aparici-Robles F, García-Gómez JM. Improving the estimation of prognosis for glioblastoma patients by MR based hemodynamic tissue signatures. NMR Biomed. 2018;31(12):e4006.
- Oltra-Sastre M, Fuster-Garcia E, Juan-Albarracin J et al. Multi-parametric MR imaging biomarkers associated to clinical outcomes in gliomas: a systematic review. Curr Med Imaging Rev. 2019;15(10):933-947.
- Dubois RW. Is the real-world evidence or hypothesis: a tale of two retrospective studies. J Comp Eff Res. 2015;4:199-201
- Sherman RE, Anderson SA, Dal Pan GJ et al. Real-World Evidence – what is it and what can it tell us? N Engl J Med. 2016;375(23):2293-2297.
- Hernán MA. Does water kill? A call for less casual causal inferences. Ann Epidemiol. 2016;26(10):674-680.
- 2019 Tutorials – OMOP Common Data Model and Standardized Vocabularies https://www.ohdsi.org/2019-tutorials-omop-common-data-model-and-standardized-vocabularies (acceso 29 de abril de 2021).
- McDevitt J, et al. Proposed CORE national Cancer Dataset: National Cancer Registry. National Cancer Registry; Ireland: 2013
- Thiese MS. Observational and interventional study design types; an overview. Biochem Med (Zagreb). 2014;24(2):199-210.
- Veiga de Cabo J, Fuente Díez E, Zimmermann Verdejo M. Modelos de estudios en investigación aplicada: conceptos y criterios para el diseño. Med Segur Trb (2008) V54.n210.
- Von Elm E, et al. The Strengthening the reporting of observational studies in epidemiology (STROBE) statement. Ann Int Med 2007; 147:573-77
- Hernán MA, Robins JM. Using Big Data to emulate a target trial when a randomized trial is not available. Am J Epidemiol. 2016; 183:758-64
- https://www.equator-network.org/ (acceso 29 de abril de 2021).
- www.chaimeleon.eu (acceso 29 de abril de 2021).
- Martí-Bonmatí L. Evidence levels in radiology: the insights into imaging approach. Insights Imaging. 2021; 12(1):45.
DECLARACIÓN DE TRANSPARENCIA
El autor/a de este artículo declara no tener ningún tipo de conflicto de intereses respecto a lo expuesto en el presente trabajo.
ranm tv
Luis Martí Bonmatí
Real Academia Nacional de Medicina de España
C/ Arrieta, 12 · 28013 Madrid
Tlf.: +34 91 547 03 18 | E-Mail: marti_lui@gva.es
Año 2021 · número 138 (01) · páginas 16 a 23
Enviado*: 23.03.21
Revisado: 30.03.21
Aceptado: 22.04.21
* Fecha de lectura en la RANM


