Resumen
Objetivos: Se analiza la evolución de la sensibilidad a diferentes antibióticos, incluyendo daptomicina, de los aislados de Enterococcus faecium y Enterococcus faecalis, junto con el consumo de este antibiótico y una revisión sistemática.
Métodos: Se revisan los trabajos publicados antes de 2022, sobre los perfiles genéticos de resistencia de los enterococos productores de infecciones, empleando la base de datos MEDLINE. Se describe la sensibilidad de los enterococos urinarios, entre 2012 y 2021. Se recogió la información del consumo de daptomicina. Se analizó su relación con la no susceptibilidad, y si la existencia de esta alteraba los perfiles de sensibilidad a otros antibióticos analizados.
Resultados: Existe un grupo heterogéneo de genes que se ven alterados cuando se produce la no susceptibilidad a daptomicina, como los sistemas LiaFSR y YycFGHIJ y las enzimas Cls y GdpD, principalmente implicados en la respuesta al estrés en la membrana celular y en el control de la concentración de fosfolípidos en la membrana. Se demuestra que, en el caso de E. faecium, hay una correlación positiva entre el consumo de daptomicina y el aumento de aislados no susceptibles, de la misma manera que la no susceptibilidad a daptomicina se asocia con alteraciones en el perfil de sensibilidad de los aislados para algunos antibióticos, tanto para E. faecium, como para E. faecalis.
Conclusión: Tras la revisión sistemática realizada se concluye que existen genes responsables de la falta de susceptibilidad a daptomicina. Hay una correlación positiva entre el uso de daptomicina y la no susceptibilidad: a mayor uso, mayor no susceptibilidad al antibiótico. Los perfiles de sensibilidad a otros antibióticos se ven asociados a la no susceptibilidad a daptomicina.Abstract
Background: It analyzes the evolution of the susceptibility profile to different antibiotics, including daptomycin, of isolates of Enterococcus faecium and Enterococcus faecalis from urinary cultures, together with the consumption of this antibiotic and a systematic review.
Methods: Studies published before 2022 on the genetic profiles of resistance of infection-producing enterococci are reviewed, using the MEDLINE database. Urinary enterococci are described between 2012 and 2021. Information on daptomycin consumption was collected. Its relationship with non-susceptibility was analyzed, and whether the existence of this altered the susceptibility profiles to other antibiotics analyzed.
Results: There is a heterogeneous group of genes that are altered when non-susceptibility to daptomycin occurs, such as the LiaFSR and YycFGHIJ systems and the enzymes Cls and GdpD, mainly involved in the response to stress in the cell membrane and in the control of the concentration of phospholipids in the membrane. It is shown that, in the case of E. faecium, there is a positive correlation between the consumption of daptomycin and the increase in non-susceptible isolates, in the same way that non-susceptibility to daptomycin is associated with alterations in the susceptibility profile of the isolates for some antibiotics, both for E. faecium and for E. faecalis.
Conclusion: After the systematic review carried out, it is concluded that there are genes responsible for the lack of susceptibility to daptomycin. There is a positive correlation between the use of daptomycin and non-susceptibility, the greater the use, the greater the non-susceptibility to the antibiotic. Sensitivity profiles to other antibiotics are associated with non-susceptibility to daptomycin.Palabras clave: Daptomicina; Sensibilidad; Resistencia; Enterococos.
Keywords: Daptomycin; Susceptibility; Resistance; Enterococci.
Introducción
Daptomicina (DAP) fue descubierta a finales de los ochenta en aislados de Streptomyces roseosporus procedentes del suelo de Turquía (1). Las máximas concentraciones séricas alcanzadas son de 160 mg/L y la semivida oscila entre 8 y 9 h, aumentando en la insuficiencia renal (2). La excreción renal es de forma activa en un porcentaje del 78% (3), por lo tanto, susceptible de ser empleado en la infección urinaria de vías altas. Su volumen de distribución es bajo (0,1 L/kg) y se limita al plasma y el líquido intersticial, con un metabolismo que todavía no es bien conocido. Es un bactericida potente y con un efecto constante, sobre todo a nivel del mesosoma septal, lugar donde se acumula cardiolipina bacteriana responsable de parte de las funciones de la membrana citoplasmática (4). Al no producir una lisis bacteriana directa, evita la liberación de citoquinas proinflamatorias. DAP muestra efecto posantibiótico, lo que permite utilizar una dosis diaria, y su efecto clínico es concentración-dependiente, dónde el valor de PK/PD y la dosis terapéutica deben adaptarse bien a la concentración mínima inhibitoria (CMI), aunque esta es difícil de calcular, para conocer el valor de la ƒÁrea bajo la curva / CMI, marcador empleado para vigilar su eficacia clínica (1). El método de referencia es la dilución, aunque su formato automatizado puede mostrar un valor ligeramente más alto y la prueba de difusión con disco no está aprobada (5). Muestra actividad in-vitro sólo frente a bacterias grampositivas, aerobios y anaerobios. Su uso en Europa está aceptado desde 2003, en pacientes mayores de un año, con infecciones graves por Staphylococcus y Streptococcus β-hemolíticos, que son resistentes a otros antibióticos (6). En el caso de Enterococcus, no existe suficiente experiencia clínica y su uso no está aprobado para tratar infecciones graves por aislados resistentes a vancomicina, a diferencia de lo que ocurre en los EE. UU.. Aquí se emplean, para Enterococcus faecalis, dosis de 6 mg/kg/día, que puede llegar hasta 12 mg/kg/día en caso de ser necesario; siendo esta dosis la empleada para Enterococcus faecium en los aislados que son “sensible dosis dependientes” (7), aunque los fracasos clínicos se han descrito incluso con CMI de 2 y 4 mg/L, por el desarrollo de resistencia o tolerancia por mutaciones de los genes del sistema LiaFSR, encargado de la reparación de la membrana celular bacteriana (5). Por eso la FDA no aprueba este fármaco para tratar las infecciones por E. faecium. Lo que sí se ha demostrado es un aumento del consumo en España de DAP (8) para diferentes escenarios clínicos, y, tanto de forma empírica como dirigida. Recientemente se informa cada vez más la aparición de resistencia a DAP en enterococos durante el tratamiento, o como un fenómeno acompañante en el tratamiento de infecciones por otros patógenos (9, 10), aunque esto no evita la aparición de aislados clínicos resistentes en sujetos sin evidencia de tratamiento previo con DAP (11, 12). A pesar de estos hechos aislados, no existen estudios globales recientes de los mecanismos de resistencia de los enterococos a DAP y sobre la evolución de la susceptibilidad en los aislamientos clínicos. Las escasas publicaciones que existen son de estudios transversales (13). En este trabajo se realiza una revisión sistemática de las publicaciones sobre los mecanismos de resistencia y se analiza la evolución de la CMI a DAP entre los aislados clínicos de Enterococcus, relacionándola con su consumo hospitalario.
Material y métodos
Revisión sistemática
Se han seguido las normas de PRISMA (https://prisma-statement.org//) para realizar esta revisión. Para la realización de la revisión sistemática sobre la base genética conocida de la resistencia a DAP en las especies E. faecium y E. faecalis se realizó una búsqueda en la base de datos MEDLINE, a través de PubMed, a fecha de 1 de diciembre de 2021, utilizando las palabras clave “Enterococcus”, “daptomycin”, y “resistant”, con un resultado total de 680 artículos. Dado que un objetivo del trabajo era el estudio de las bases genéticas de la resistencia a DAP, se seleccionaron aquellos que trataran esas bases de manera demostrable dando como resultado genes concretos. Se elaboró una tabla con la siguiente información: la especie microbiana estudiada, la metodología utilizada para obtener los resultados, el gen que se encontró modificado y, en los casos posibles, la posición y tipo de mutación que produce la resistencia a DAP.
Análisis de la evolución de la CMI a DAP y su consumo hospitalario.
Se realizó un análisis del perfil de sensibilidad de los Enterococcus aislados en todos los urocultivos significativos en el Laboratorio de Microbiología del Hospital Universitario Virgen de las Nieves (HUVN) de Granada entre el 1 de enero 2012 y el 31 de diciembre de 2021. El Hospital es un complejo asistencial regional constituido por tres centros (Hospital General de Especialidades, Hospital Materno-Infantil y Hospital de Neuro-Traumatología y Rehabilitación) que tiene una actividad asistencial de tercer nivel en la provincia. En nuestro estudio, la población asistida estuvo constituida por pacientes procedentes exclusivamente de atención especializada, como signo de importancia clínica. Se careció de la información expresa sobre los síntomas urinarios. Las muestras, obtenidas mediante sondaje provisional, micción media, sonda permanente, catéter de nefrostomía y bolsa colectora de niños, fueron procesadas siguiendo un protocolo estricto de trabajo (14). Una vez clasificadas las muestras se procedió a la identificación de las colonias y estudio sensibilidad a antimicrobianos mediante microdilución automatizada (MicroScan Walkaway, Beckman Coulter, Barcelona, España). Los antibióticos ensayados fueron DAP, ampicilina, ciprofloxacino, eritromicina, estreptomicina (512 mg/L), fosfomicina, gentamicina (128 mg/L), levofloxacino, linezolid, nitrofurantoina, penicilina, synercid para E. faecium, tetraciclina, teicoplanina y vancomicina. Los aislados se clasificaron en sensibles, intermedios o resistentes a cada antibiótico de acuerdo con las recomendaciones del European Committee on Antimicrobial Susceptibility Testing (EUCAST; https://www.eucast.org/) del año 2022, y para DAP se estratificaron los resultados en < 1, 2, 4 y > 4mg/L. Se usaron criterios del CLSI 2022 en el caso de penicilina, eritromicina, tetraciclina y fosfomicina, con igual criterio en ambas especies. Una vez realizados los urocultivos se realizó un estudio descriptivo-retrospectivo, partiendo de todos los datos y resultados registrados en el sistema de información del laboratorio (SIL) del Servicio de Microbiología del HUVN, para calcular los porcentajes de cada categoría clínica de cada antibiótico. No se excluyeron del estudio las muestras de pacientes con factores de riesgo tales como tratamiento inmunosupresor, cirugía urológica reciente, neovejiga, u otro tipo de derivación urinaria, vejiga neurógena, procedimientos invasivos sobre la vía urinaria y antecedentes de profilaxis antibiótica. Se excluyeron los resultados procedentes de muestras diferentes de un mismo paciente cuando, con igual resultado microbiológico, fueron obtenidas con un intervalo inferior a 20 días; así como cuando la muestra clínica no estuviera correctamente filiada o el volante de petición analítica no estuviera correctamente relleno.
Además, para conocer la capacidad de la prueba de gradiente de difusión para detectar la CMI de DAP, en una muestra de 98 aislados clínicos de 2020 y 2021 que presentaron una CMI >4 mg/L con MicroScan (14-14,28%- E. faecalis y 84-85,72%- E. faecium) fueron estudiados mediante E-test (Liofilchem, Rosetodegli Abruzzi, Italia) en medio sólido de Mueller-Hinton (BD, Madrid, España), con lectura a las 24h. de incubación a 37ºC.
Análisis estadístico
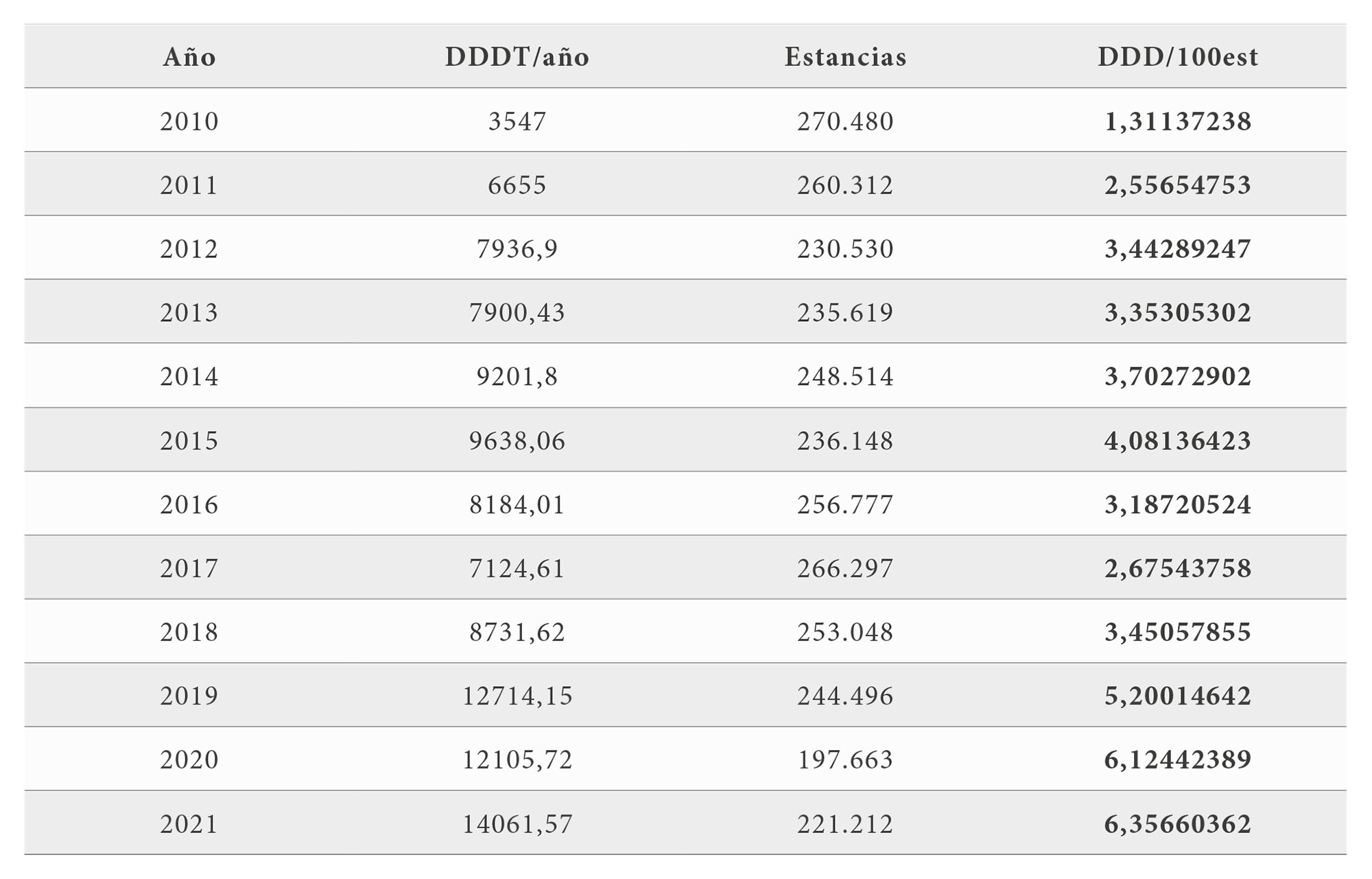
Para estudiar la relación entre la susceptibilidad a daptomicina y el resto de los antibióticos estudiados, tanto para E. faecium como E. faecalis, se realizaron tablas de contingencia, y se aplicaron las pruebas chi-cuadrado de Pearson o exacto de Fisher, en los casos con más del 20% de las frecuencias esperadas inferiores a 5. Tras calcular el consumo anual de DAP por cada 100 estancias (DDD/1000 stays), se estudió la relación entre éste y el aumento de la CMI, mediante el coeficiente de correlación de Pearson para E. faecium y el coeficiente de correlación de Spearman para E. faecalis, debido al incumplimiento de la hipótesis de normalidad. La normalidad de las variables numéricas se contrastó con la prueba de Shapiro-Wilks. Para todos los contrastes se consideró un nivel de significación de 0,05. Los datos se analizaron con IBM SPSS Statistics 19 y R 4.0.5.
El protocolo de estudio se realizó conforme con la Declaración de Helsinki. Durante éste, no se realizó ninguna investigación adicional a los procedimientos rutinarios, y se utilizó solo el material biológico para el diagnóstico estándar de infecciones urinarias prescrito por los médicos. Se realizaron los análisis de datos utilizando una base de datos anónima, donde los sujetos fueron identificados sólo mediante el Número Único de Historia de Salud de Andalucía (NUHSA) y sustituidos por episodios infecciosos diferentes. La entidad que concedió el permiso para acceder a los datos y utilizarlos fue la Unidad de Gestión Clínica de Microbiología Clínica del Hospital Virgen de las Nieves de Granada, España.
Resultados
Revisión Sistemática
Tras realizar la revisión sistemática de los trabajos publicados se seleccionaron 14 publicaciones (tabla 1) que estudiaron las bases genéticas de la resistencia a DAP de manera demostrable, dando como resultado genes concretos: 6 trabajos estudiaron las resistencias en E. faecium, 5 en E. faecalis y 3 en ambas especies. La metodología de estos estudios es principalmente secuenciación Illumina y en algunos casos mutagénesis, aunque también utilizan otras metodologías como secuenciación Sanger o análisis de MLST, pero con menor frecuencia. El grupo de genes más estudiado son los pertenecientes a la familia del sistema LiaFSR, remarcando LiaS y LiaR. En estos genes se encontraron mutaciones en sitios concretos de la proteína final de la transcripción de estos, en el caso de LiaS resaltó la sustitución T120S y en el caso de LiaR la sustitución W73C (15, 16, 17, 18), aunque en LiaR también encontramos sustitución en S19F (11). También encontramos el gen LiaX con deleción en 289 (19). Por último, en este grupo encontramos que las mutaciones en LiaF también son importantes para el fenotipo resistente, marcando en LiaF la deleción 177 (19, 20, 21). Por otro lado, hay trabajos que estudian los genes que codifican para Cls u GdpD, que son enzimas que regulan la concentración de fosfolípidos en la membrana. En este caso la concordancia en el sitio de mutación no es tan grande, sino que podemos observar cómo mutaciones diferentes conllevan al fenotipo resistente. En Cls encontramos las sustituciones H215R (22, 23), R267C (24), P181H (24), R218Q (25); deleción en 61 (18); y MPL110 (18). En el caso de GdpD, aunque se estudia en más trabajos, sólo tenemos mutación detectada en uno, la deleción en 170 (19, 26). También participan en el fenotipo de resistencia o tolerancia las proteínas codificas en el sistema YycFG y su sistema de proteínas accesorias YycHIJ. Las mutaciones en estos genes por sí solas no la producen, sino que colaboran en la aparición y aunque se nombra en varios estudios solo aparece una mutación detectada, la sustitución S333L en el gen YycG (19, 20).
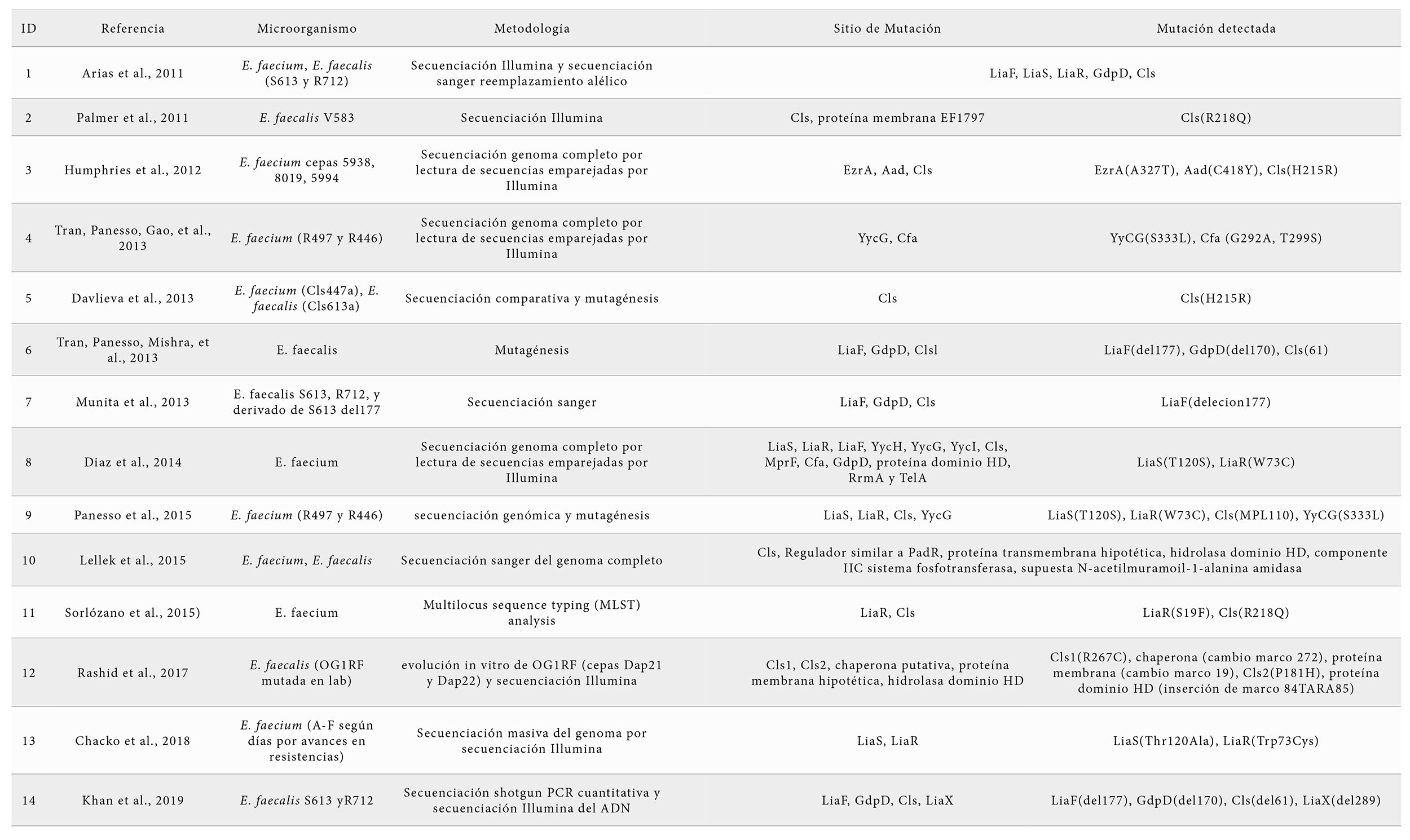
Se han descrito otros mecanismos menos importantes, pero que también participan en la regulación de los fosfolípidos de membrana o en la respuesta al estrés y que pueden contribuir a la aparición de resistencias a DAP. Así, se describe la implicación de las sustituciones G292A y T299S en el gen Cfa (26), alteraciones en los genes MprF, RrmA o TelA (15), sustitución A327T en el gen EzrA y sustitución C418Y en el gen Aad (22), y cambios en el componente IIC del sistema fosfotransferasa o en la N-acetilmuramoil-1-alanina amidasa (27).
Análisis de la evolución de la CMI a DAP y su consumo hospitalario
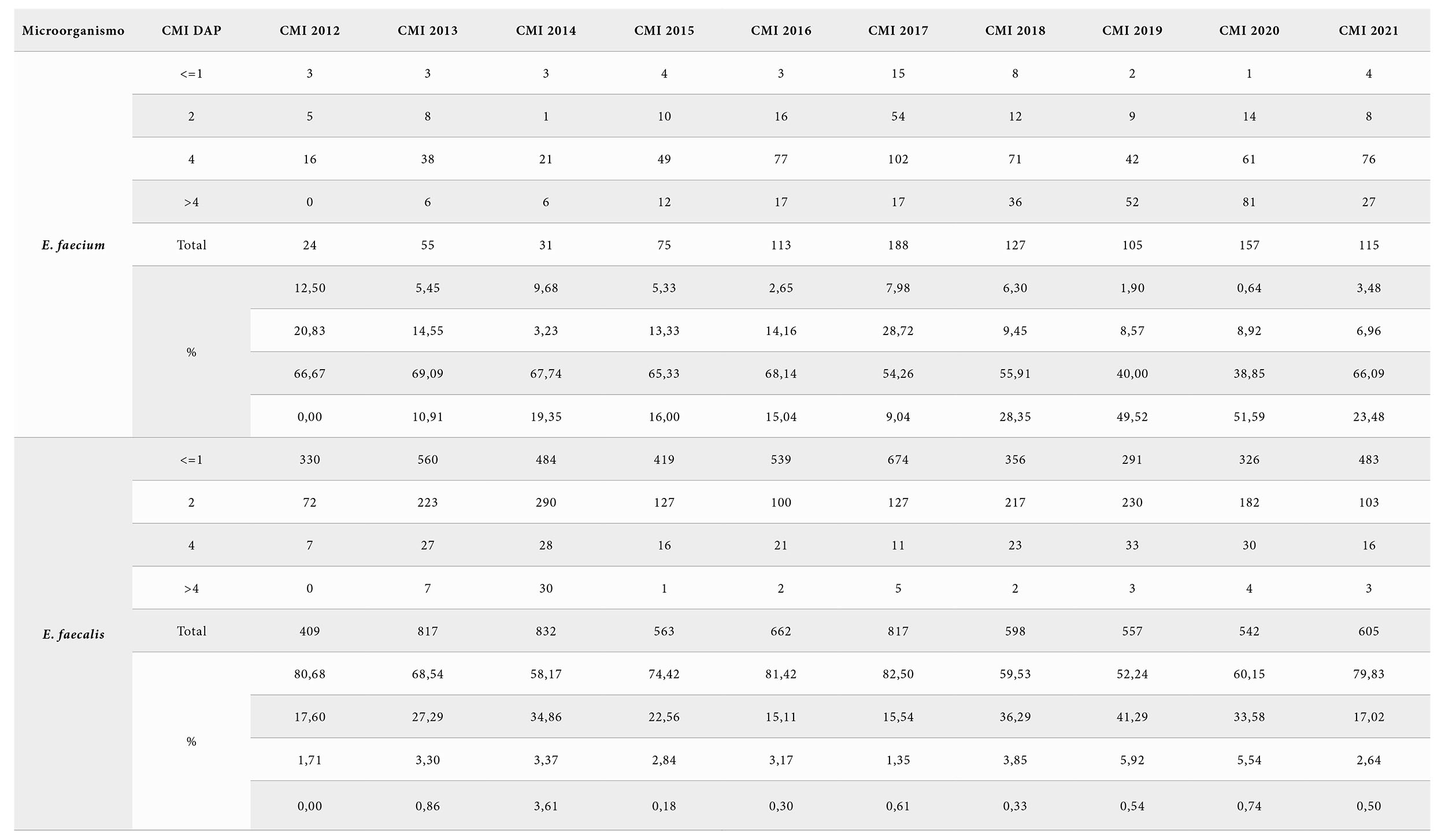
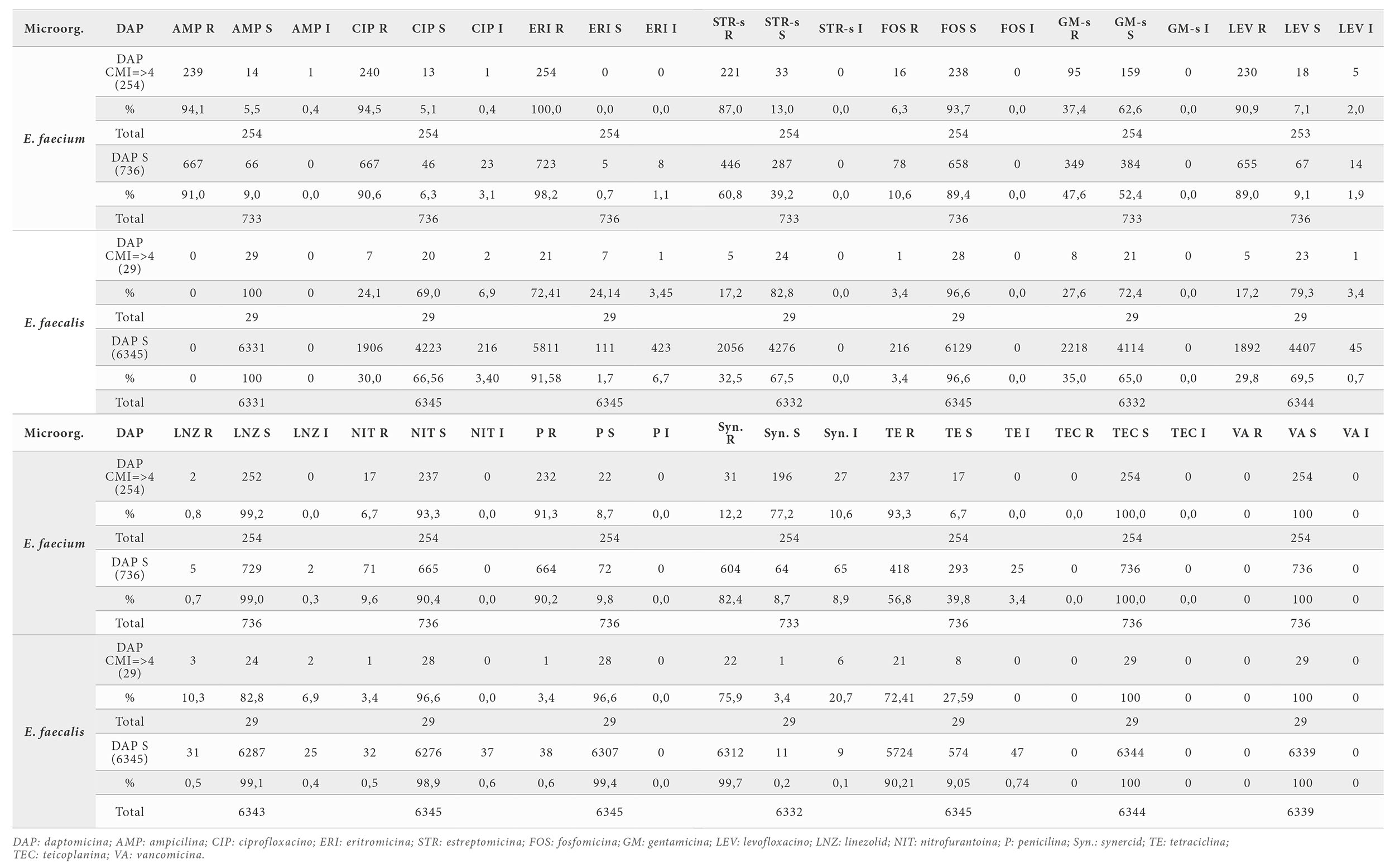
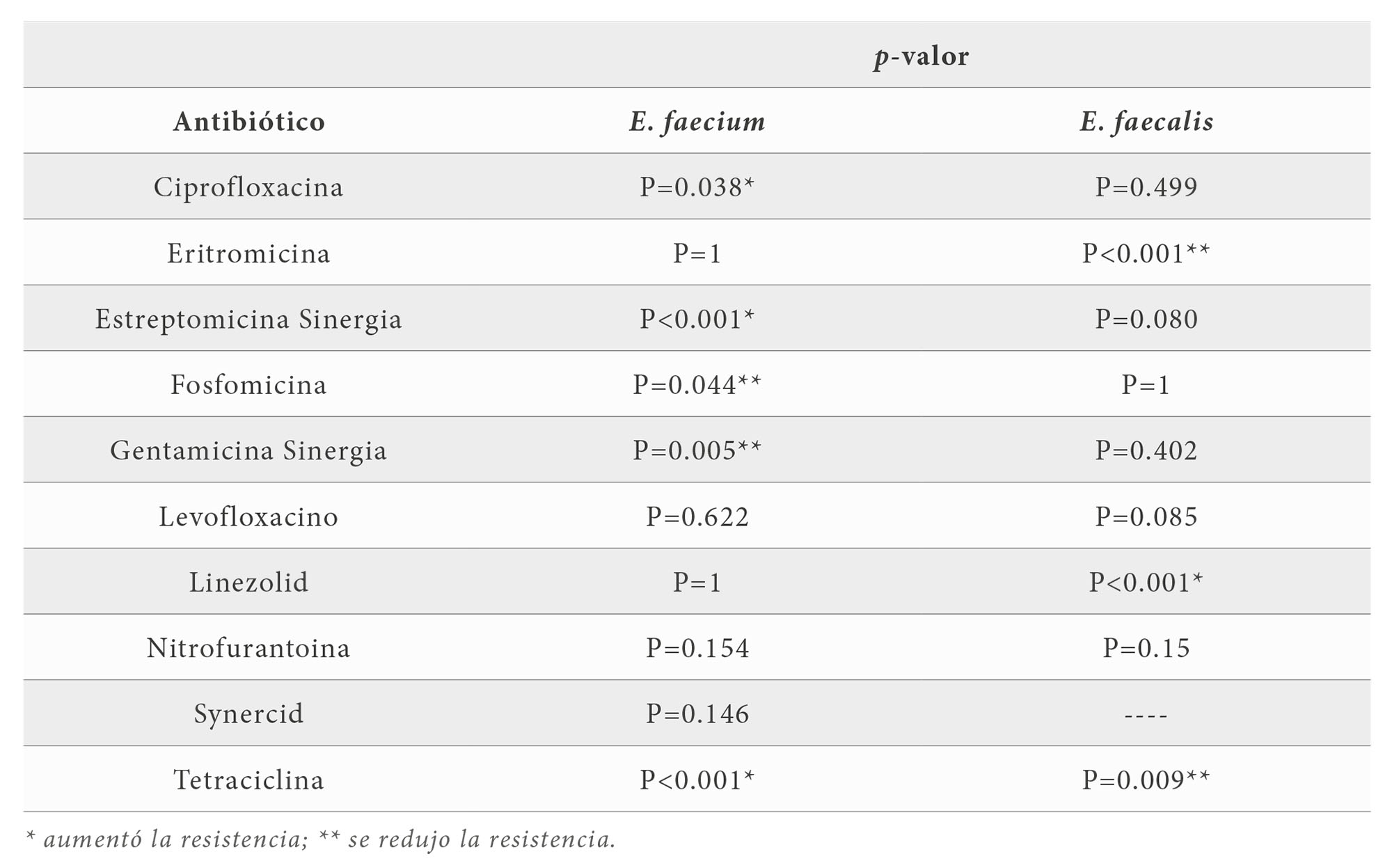
Se estudiaron 79.913 urocultivos, de los que resultaron positivos 29.568 (37%) y de los cuales Enterococcus fueron 7.392 (25%): 990 (13,4%) E. faecium y 6.402 (86,6%) E. faecalis. Se obtuvo la información sobre la susceptibilidad a 15 antibióticos (tablas 2 y 3), destacando un incremento anual de los aislados de E. faecium con una CMI > 4 mg/L de DAP. En la tabla 4 se muestra el consumo de DAP por año, y los datos calculados por cada 100 estancias por año. Para los aislados clínicos de E. faecium existe una correlación negativa estadísticamente significativa (p=0,018; p=0,02), entre el consumo anual y consumo anual por cada 100 estancias de DAP, y el porcentaje de aislados clínicos con CMI < 4 mg/L (figura 1): cuanto mayor es el consumo anual de DAP menor es el porcentaje de aislados clínicos con CMI < 4 mg/L. En el caso de los aislados de E. faecalis, no se puede demostrar, de momento, que exista una correlación estadísticamente significativa (p=0,987; p=0,855) entre el consumo anual y consumo anual por cada 100 estancias de DAP, y el porcentaje de aislados cuyo CMI < 4 mg/L. En la tabla 5 se relaciona la resistencia al resto de los antibióticos y los valores de CMI > 4mg/L de DAP. De algunos antibióticos, como ampicilina, vancomicina y teicoplanina, no se pudo calcular el estadístico debido a que todos los aislados tuvieron igual comportamiento. Así los antibióticos para los que hubo asociación entre su fenotipo de resistencia, con aumento o reducción de esta, y la CMI > 4 mg/L de DAP fueron, en E. faecium, para ciprofloxacino (p= 0,038), estreptomicina sinergia (p<0,001), fosfomicina (p=0,044), gentamicina sinergia (p=0,005) y tetraciclina (p<0,001). En el caso de E. faecalis se encontró asociación en eritromicina (p<0,001), linezolid (p<0,001), y tetraciclina (p<0,009). En el caso de E. faecium, aumentó la resistencia a ciprofloxacina, tetraciclina y estreptomicina, y se redujo a fosfomicina y gentamicina. En el caso de E. faecalis, aumentó la resistencia a linezolid y se redujo eritromicina y tetraciclina. Con la prueba del gradiente de difusión sólo 35 (35,7%) aislados de E. faecium, de los 98 enterococos estudiados, tuvieron una CMI > 4 mg/L.
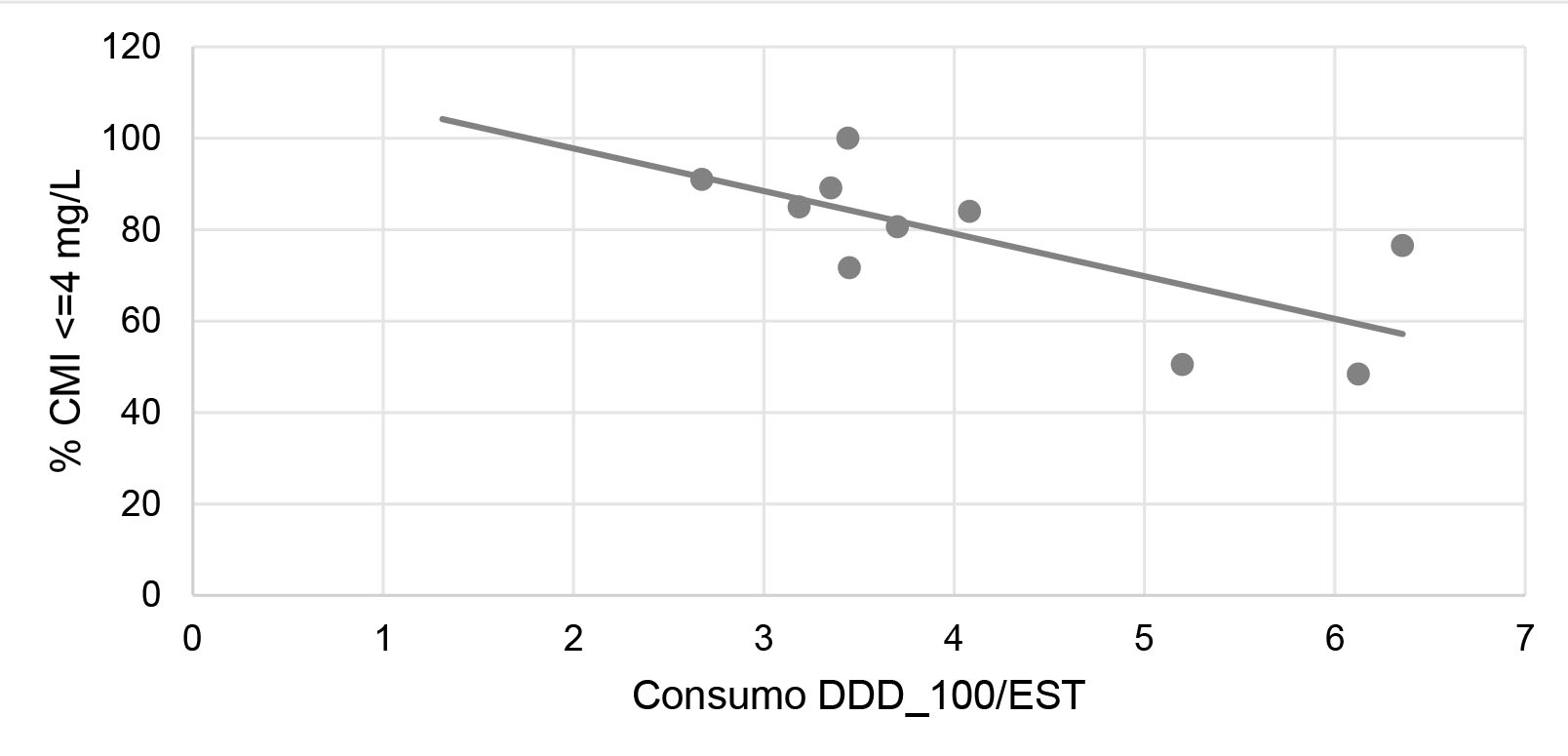
% anual de E. faecium con CMI < 4 mg/L: 2012 = 100%; 2013 = 89,09%; 2014 = 80,65%; 2015 = 84%; 2016 = 84,96%; 2017 = 90,96%; 2018 = 71,65%; 2019 = 50,48%; 2020 = 48,41%; 2021 = 76,52%.
Discusión
Aunque DAP ha sido una incorporación útil al repertorio de antibióticos a usar, según su uso clínico avanza, los informes de resistencias entre los enterococos son más habituales. Por eso comprender mejor la biología de la adaptación a DAP es crucial para conservar su utilidad médica. En este trabajo se resalta como la CMI a DAP entre los aislamientos clínicos de E. faecium ha aumentado con el paso de los años en nuestro medio. Son muchos los genes y mecanismos descritos que han sido relacionados con la aparición de resistencia a DAP (tabla 1) y que pueden ser los responsables del aumento de esta CMI. De forma resumida estos incluyen las alteraciones de los sistemas de control del estrés en la membrana celular y sistemas del control de homeostasis, con su base en genes que se encuentran dentro de los sistemas LiaFSR y YycFGHIJ, así como genes relacionados con la concentración y el metabolismo de fosfolípidos en la membrana, la CLs y la GdpD. Las mutaciones más comunes son las de LiaR W73C, LiaS T120A, LiaF del177 y CLs H215R y R218Q. Aun así, no hay que olvidar la implicación de otros genes, como EzrAo Aad, así como la proteína hipotética de membrana o el gen de la hidrolasa de la familia HD (28). Serán necesario estudios posteriores para conocer que está pasando en nuestros aislados, que hemos analizado, con CMI incrementada a DAP, aunque sí podemos comprender a qué se puede deber.
En E. faecium, demostramos que a medida que el consumo de DAP ha aumentado, el porcentaje de aislados con CMI<4 mg/L en nuestro medio ha disminuido, como se ve en la figura 1, esto implica que a mayor consumo de DAP en nuestro medio mayor es el riesgo de que las posibles infecciones por E. faecium tengan potencial resistencia a este antibiótico. En el caso de E. faecalis, no hay información suficiente para demostrar que exista, con valor estadísticamente significativo, un aumento de los aislados con CMI > 4 mg/L a DAP. Habiendo demostrado un aumento de la CMI a DAP en E. faecium en nuestro medio, y teniendo en cuenta que de por sí ya suelen ser resistentes a ciprofloxacino, estreptomicina y tetraciclina, se deberían hacer estudios microbiológicos más detallados de cada aislamiento, para evitar seguir incrementado las tasas de resistencias por el uso incorrecto de los tratamientos empíricos. En esta resistencia múltiple en los E. faecium la no susceptibilidad a DAP puede haber influido en la resistencia a otros antibióticos o se puede simultanear otros mecanismos de resistencia independientes. En cualquiera de los casos supone un aumento global de las resistencias.
Por encima de los valores de CMI a DAP y su categorización clínica, están los datos farmacocinéticos, dónde el valor de PK/PD y la dosis terapéutica deben adaptarse bien a la CMI, para conocer el valor de la ƒÁrea bajo la curva / CMI, marcador que debe ser superior a 27,4 para conseguir un efecto bactericida y buena supervivencia a los 30 días en la bacteriemia enterocócica (29), pero las dosis aprobadas por la EMA son de hasta 12 mg/Kg/día (30) y con estas dosis sólo se consigue que este valor del marcador cuando la CMI es < 2 mg/L Por lo tanto, la mayor parte de nuestros E. faecium no serían destruidos con estas dosis. Sería necesario comprobar si es posible usar dosis más elevadas y cuáles son las concentraciones alcanzables con estas.
Otro asunto importante es que la mayor parte de los laboratorios de microbiología sólo disponen de pruebas de microdilución automatizada que a veces aportan valores de CMI una dilución por encima de los reales, según se ha descrito (5). Una alternativa es el uso de pruebas de gradiente de difusión (E-test) que en nuestras manos no aporta suficiente sensibilidad para detectar valores de CMI > 4 mg/L. Esto se puede deber a la dificultad de DAP para difundir en el agar, como ocurre con vancomicina (9). Algunos autores recomiendan el uso de E-test en BHIA, ya que permite detectar los aislados con CMI elevada debido a mutaciones en el sistema liaFSR (31). Pero no se puede olvidar que este método no es un método estandarizado y, conceptualmente, no permite tomarlo como referencia para evaluar los resultados de otros métodos (como sí permite la microdilución de referencia, perfectamente realizable para un estudio de investigación). Consecuentemente, no se dispone de un método fiable para detectar los aislados reales con CMI > 4 mg/L.
Una limitación de nuestro trabajo es que para evaluar la relación entre la sensibilidad a DAP y a otros antimicrobianos, hubiera sido deseable estudiar la relación clonal de los aislados de Enterococcus (al menos de los no sensibles), pues podría haberse producido una expansión de uno o unos pocos clones que expliquen los resultados obtenidos. Otra limitación es el reducido número de artículos analizados, solo 14, lo que de alguna forma limita los resultados y conclusiones obtenidos.
En cualquiera de los casos, en nuestro medio es cierto que se ha producido un aumento de la CMI a DAP con el paso de los años, que coincide con el aumento del consumo y con la pandemia por COVID durante los años 2020 y 2021. Consecuentemente, se ha perdido la posibilidad de tratar con DAP las infecciones por aislados con CMI más bajas. No obstante, se sabe que DAP tiene similitud estructural con el grupo de moléculas producidas por el sistema inmunitario inespecífico de los mamíferos, conocido como “péptidos catiónicos antimicrobianos”, como la catelicidina humana LL-37 (5) y es posible que lo que estemos detectando es la respuesta de este microorganismo a este grupo de sustancias.
En conclusión, en nuestro medio, a lo largo de los años, se ha producido un aumento de los aislados clínicos de E. faecium no susceptibles a DAP, que puede ser debido al aumento del consumo de este en el medio clínico, pero que siguen siendo sensibles a fosfomicina y gentamicina.
DECLARACIÓN DE TRANSPARENCIA
Los autores/as de este artículo declaran no tener ningún tipo de conflicto de intereses respecto a lo expuesto en el presente trabajo.
BIBIOGRAFÍA
- ↑Karas JA, Carter GP, Howden BP et al. Structure-Activity Relationships of Daptomycin Lipopeptides. J Med Chem. 2020; 63(22): 13266-13290. https://pubs.acs.org/doi/10.1021/acs.jmedchem.0c00780
- ↑Heidary M, Khosravi AD, Khoshnood S, Nasiri MJ, Soleimani S, Goudarzi M. Daptomycin. J AntimicrobChemother. 2018; 73(1): 1-11. https://doi.org/10.1093/jac/dkx349
- ↑Beiras-Fernandez A, Vogt F, Sodian R, Weis F. Daptomycin: a novel lipopeptide antibiotic against Gram-positive pathogens. Infect Drug Resist. 2010; 3: 95-101. https://doi.org/10.2147/IDR.S6961
- ↑Turner AM, Lee JYH, Gorrie CL, Howden BP, Carter GP. Genomic Insights Into Last-Line Antimicrobial Resistance in Multidrug-Resistant Staphylococcus and Vancomycin-Resistant Enterococcus. Front Microbiol. 2021; 12: 637-656. https://doi.org/10.3389/fmicb.2021.637656
- ↑Khan A, Miller WR, Axell-House D, Munita JM, Arias CA.AntimicrobialSusceptibilityTestingforEnterococci. J Clin Microbiol. 2022; 60(9): e0084321. https://doi.org/10.1128/jcm.00843-21
- ↑Vidal P, Fourniols E, Junot H, Meloni C, Bleibtreu A, Aubry A. Antibiotic Stewardship in Treatment of Osteoarticular Infections Based on Local Epidemiology and Bacterial Growth Times. MicrobiolSpectr.: e0143022. https://doi.org/10.1128/spectrum.01430-22
- ↑Clinical and Laboratory Standards Institute (CLSI). Performance Standards for Antimicrobial Susceptibility Testing. 31st ed. CLSI supplement M100 (ISBN 978-1-68440-104-8 [Print]; ISBN 978-1-68440-105-5 [Electronic]). Clinical and Laboratory Standards Institute, USA, 2021. (n.d.). Retrieved August 30, 2022, from https://clsi.org/standards/products/microbiology/documents/m100.
- ↑Del Rosario-García B, Nazco-Casariego GJ, Gómez-Sirvent JL et al. Vancomycin versus daptomycin for the treatment of confirmed gram-positive catheter-related bloodstream infections in oncology patients. Farm Hosp. 2022; 46(3): 105-108 https://dx.doi.org/10.7399/fh.11724
- ↑Turnidge J, Kahlmeter G, Cantón R, MacGowan A, Giske CG; European Committee on Antimicrobial Susceptibility Testing. Daptomycin in the treatment of enterococcal bloodstream infections and endocarditis: a EUCAST position paper. Clin Microbiol Infect. 2020; 26(8): 1039-1043. https://doi.org/10.1016/j.cmi.2020.04.027
- ↑Zhou, X, Willems, RJL, Friedrich, AW, Rossen JWA, Bathoorn E. Enterococcus faecium: from microbiological insights to practical recommendations for infection control and diagnostics. Antimicrob Resist Infect Control. 2020; 9, 130. https://doi.org/10.1186/s13756-020-00770-1
- ↑Sorlózano A, Panesso D, Navarro-Marí JM, Arias CA, Gutiérrez-Fernández J. Characterization of daptomycin non-susceptible Enterococcus faecium producing urinary tract infection in a renal transplant recipient. Rev Esp Quimioter. 2015; 28(4): 207-9. PMID: 26200029; PMCID: PMC5668868
- ↑Kelesidis T, Humphries R, Uslan DZ, Pegues D. De novodaptomycin-nonsusceptibleenterococcalinfections. Emerg Infect Dis. 2012; 18(4): 674-6. http://dx.doi.org/10.3201/eid1804.110932
- ↑DiPippo AJ, Tverdek FP, Tarrand JJ et al. Daptomycin non-susceptible Enterococcus faecium in leukemia patients: Role of prior daptomycin exposure. J Infect. 2017; 74(3): 243-247. doi: 10.1016/j.jinf.2016.11.
- ↑Rodríguez Del Águila MM, Sorlózano-Puerto A, Fernández-Sierra MA, Navarro Marí JM, Gutiérrez Fernández J. Sociodemographic characteristics and risk factors associated to significative bacteriuria in a Spanish health area. Rev Esp Quimioter. 2022; 35(4): 382-391. Spanish. doi: 10.37201/req/016.2022. https://doi.org/10.1056/NEJMOA1011138
- ↑Diaz L, Tran TT, Munita JM et al. Whole-genome analyses of Enterococcus faecium isolates with diverse daptomycin MICs. Antimicrobial Agents and Chemotherapy. 2014;58(8), 4527–4534. https://doi.org/10.1128/AAC.02686-14
- ↑Panesso D, Reyes J, Gaston EP et al. Deletion of liaR Reverses Daptomycin Resistance in Enterococcus faecium Independent of the Genetic Background. Antimicrob Agents Chemother. 2015;59(12), 7327–7334. https://doi.org/10.1128/AAC.01073-15.
- ↑Chacko KI, Sullivan MJ, Beckford C et al. Genetic Basis of Emerging Vancomycin, Linezolid, and Daptomycin Heteroresistance in a Case of Persistent Enterococcus faecium Bacteremia. Antimicrob Agents Chemother. 2018;62(4). https://doi.org/10.1128/AAC.02007-17
- ↑Arias CA, Panesso D, McGrath DM et al. Genetic basis for in vivo daptomycin resistance in enterococci. New Engl J of Med. 2011; 365(10): 892-900. https://doi.org/10.1056/NEJMOA1011138
- ↑Khan A, Davlieva M, Panesso D et al. Antimicrobial sensing coupled with cell membrane remodeling mediates antibiotic resistance and virulence in Enterococcus faecalis. Proceedings of the National Academy of Sciences of the United States of America. 2019;116(52), 26925–26932 https://doi.org/10.1073/PNAS.1916037116.
- ↑Tran TT, Panesso D, Mishra NN et al. Daptomycin-resistant Enterococcus faecalis diverts the antibiotic molecule from the division septum and remodels cell membrane phospholipids. MBio. 2013; 4(4): e00281-13. https://doi.org/10.1128/MBIO.00281-13
- ↑Munita JM, Tran TT, Diaz L et al. A liaF codon deletion abolishes daptomycin bactericidal activity against vancomycin-resistant Enterococcus faecalis. Antimicrob Agents Chemother. 2013;57(6), 2831–2833. https://doi.org/10.1128/AAC.00021-13
- ↑Humphries RM, Kelesidis T, Tewhey R et al. Genotypic and phenotypic evaluation of the evolution of high-level daptomycin nonsusceptibility in vancomycin-resistant Enterococcus faecium. Antimicrob Agents Chemother. 2012; 56(11), 6051–6053. https://doi.org/10.1128/AAC.01318-12.
- ↑Davlieva M, Zhang W, Arias CA, Shamoo Y. Biochemical characterization of cardiolipin synthase mutations associated with daptomycin resistance in enterococci. Antimicrob Agents Chemother. 2013;57(1), 289–296. https://doi.org/10.1128/AAC.01743-12
- ↑Rashid R, Cazenave-Gassiot A, Gao IH et al. Comprehensive analysis of phospholipids and glycolipids in the opportunistic pathogen Enterococcus faecalis. PloS One. 2017;12(4) https://doi.org/10.1371/JOURNAL.PONE.0175886
- ↑Palmer KL, Daniel A, Hardy C, Silverman J, Gilmore MS. Genetic basis for daptomycin resistance in enterococci. Antimicrob Agents Chemother. 2011;55(7): 3345–3356. https://doi.org/10.1128/AAC.00207-11
- ↑Tran TT, Panesso D, Gao H et al. Whole-genome analysis of a daptomycin-susceptible Enterococcus faecium strain and its daptomycin-resistant variant arising during therapy. Antimicrob Agents Chemother. 2013, 57(1), 261–268. https://doi.org/10.1128/AAC.01454-12
- ↑Lellek H, Franke GC, Ruckert C et al. Emergence of daptomycin non-susceptibility in colonizing vancomycin-resistant Enterococcus faecium isolates during daptomycin therapy. Intern J Medical Microbiology: IJMM. 2015; 305(8), 902–909. https://doi.org/10.1016/J.IJMM.2015.09.005
- ↑Reyes Manrique JC. Contribución del sistema regulatorio de tres componentes liaFSR en la resistencia a daptomicina en Enterococcusfaecalis y Enterococcusfaecium. Tesis doctoral. Bogotá. 2014. Disponible en https://repository.javeriana.edu.co/bitstream/handle/10554/15427/ReyesManriqueJinnetheCristina2014.pdf?sequence=1
- ↑Satlin MJ, Nicolau DP, Humphries RM et al. Development of Daptomycin Susceptibility Breakpoints for Enterococcus faecium and Revision of the Breakpoints for Other Enterococcal Species by the Clinical and Laboratory Standards Institute. Clinical Infectious Diseases: 2020¸ 70(6): 1240–1246. https://doi.org/10.1093/cid/ciz845
- ↑Carvajal-Fernández A, Fernández-García M, Aparicio-Hernández RM, García-Luque A. Estudio de prescripción-indicación de daptomicina en el Hospital Central de la Defensa «Gómez Ulla». Sanid. Mil. 2021; 77(4): 198-202. https://dx.doi.org/10.4321/s1887-85712021000400005
- ↑Munita JM, Panesso D, Diaz L et al. Correlation between mutations in liaFSR of Enterococcus faecium and MIC of dap2tomycin: revisiting daptomycin breakpoints. Antimicrob Agents Chemother. 2012; 56(8): 4354-9. doi: 10.1128/AAC.00509-12.
José Gutiérrez-Fernández
Laboratorio de Microbiología. Hospital Virgen de las Nieves
Av. de las Fuerzas Armadas, 2 · 18014 Granada
E-Mail: josegf@ugr.es
Año 2023 · número 140 (03) · páginas 284 a 297
Enviado: 21.10.23
Revisado: 07.11.23
Aceptado: 30.11.23


