Resumen
Los tumores neuroblásticos son las neoplasias sólidas extracraneales más comunes en la infancia, derivando de células de la cresta neural. Su diagnóstico puede variar en presentación clínica y pronóstico. La estadificación inicial y la caracterización del tumor son esenciales para la adaptación del tratamiento. Este estudio explora el uso de biomarcadores de imagen radiómica para mejorar la predicción de la agresividad tumoral. Se realizó un estudio multicéntrico retrospectivo en el seno del proyecto PRIMAGE con 508 pacientes diagnosticados de tumores neuroblásticos, usando imágenes de Resonancia Magnética (RM) procesadas mediante técnicas de preparación de la imagen y segmentación automática. Se extrajeron 107 características radiómicas y se aplicaron modelos de supervivencia, incluyendo análisis de SHAP para determinar las variables predictivas. La segmentación automática mostró alta concordancia con la manual (CSD = 0.965). Las características radiómicas fueron estables y reproducibles, con el modelo de supervivencia destacando 10 variables relevantes, entre ellas el eje menor del tumor y la asimetría de la distribución de intensidades. Estos factores se correlacionaron positivamente con el riesgo de mortalidad. Los biomarcadores de imagen radiómica mejoran la caracterización de tumores neuroblásticos y su pronóstico, contribuyendo a la superación de las limitaciones de los sistemas de estadificación actuales. La normalización estandarizada y la segmentación automática son cruciales para obtener resultados confiables. Este estudio establece un paso hacia la incorporación de características radiómicas como biomarcadores no invasivos en la práctica clínica.
Abstract
Neuroblastic tumors are the most common extracranial solid neoplasms in childhood, arising from neural crest cells. Their diagnosis can vary in clinical presentation and prognosis. Initial staging and tumor characterization are essential for treatment adaptation. This study explores the use of radiomic imaging biomarkers to enhance the prediction of tumor aggressiveness. A multicenter retrospective study was conducted as part of the PRIMAGE project, involving 508 patients diagnosed with neuroblastic tumors, utilizing processed magnetic resonance imaging (MRI) through image preparation techniques and automatic segmentation. A total of 107 radiomic features were extracted, and survival models were applied, including SHAP analysis to identify predictive variables. Automatic segmentation demonstrated high agreement with manual segmentation (CSD = 0.965). The radiomic features were stable and reproducible, with the survival model highlighting 10 relevant variables, including the tumor’s minor axis and the skewness of intensity distribution. These factors were positively correlated with mortality risk. Radiomic imaging biomarkers improve the characterization and prognosis of neuroblastic tumors, addressing the limitations of current staging systems. Standardized normalization and automatic segmentation are crucial for obtaining reliable results. This study establishes a step toward the incorporation of radiomic features as non-invasive biomarkers in clinical practice.
Palabras clave: Neuroblastoma; Resonancia Magnética; Biomarcadores de Imagen; Supervivencia; Modelos Predictivos.
Keywords: Neuroblastoma; Magnetic Resonance Imaging; Imaging Biomarkers; Survival; Predictive Models.
INTRODUCCIÓN
Los tumores neuroblásticos constituyen las neoplasias sólidas extracraneales más frecuentes en la edad pediátrica (1). Estas neoplasias derivan de células de la cresta neural, y comprenden el ganglioneuroma (benigno), el ganglioneuroblastoma (con potencial maligno) y el neuroblastoma (maligno). La mediana de edad al diagnóstico es de 16 meses. Estos tumores se presentan en una amplia variedad de ubicaciones, siendo las más comunes la región suprarrenal (48%), el retroperitoneo extraadrenal (25%) y el tórax (16%), seguidos por el cuello (3%) y la pelvis (3%) (2,3) (Figura 1).
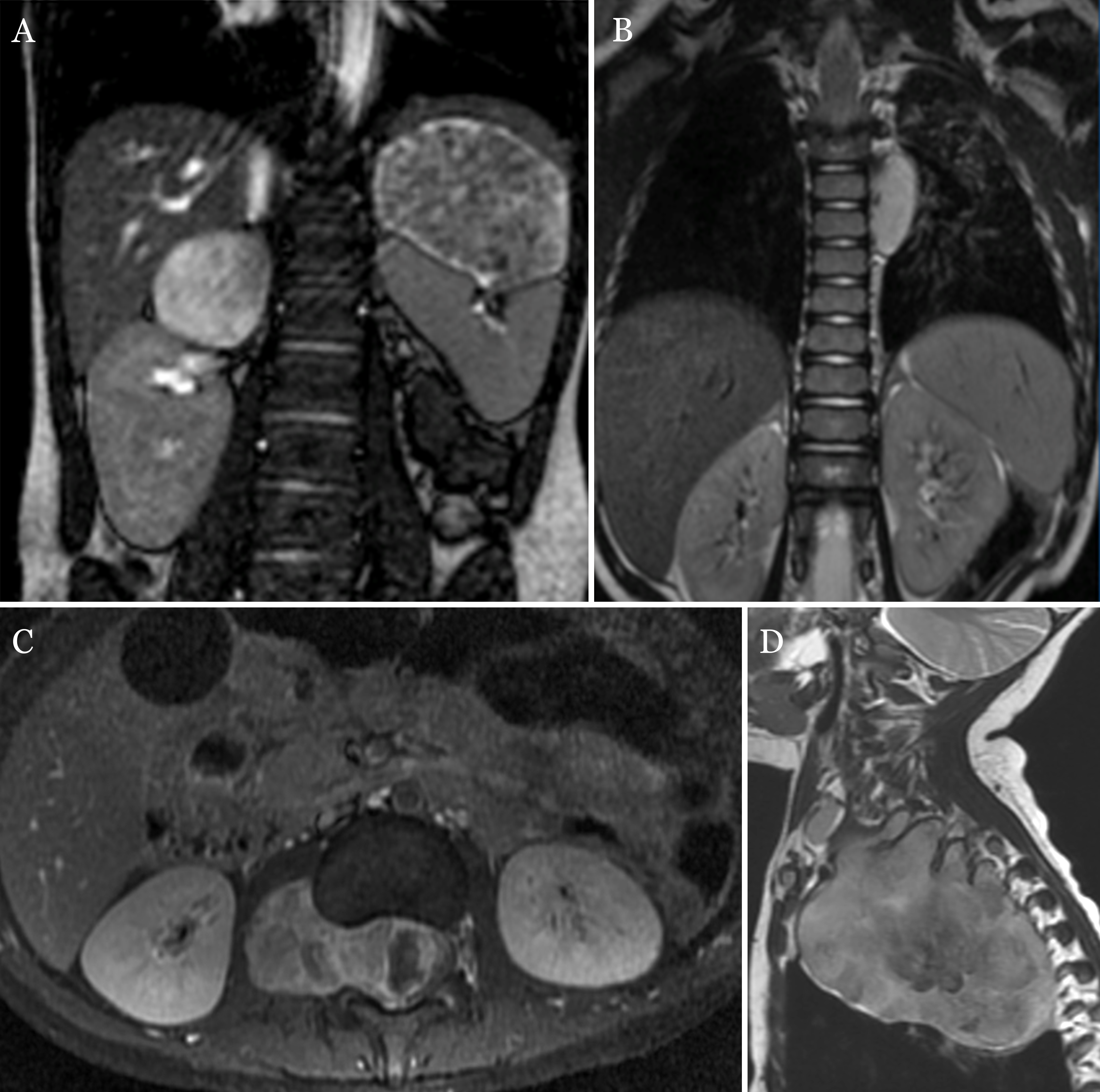
torácica izquierda con extensión al estrecho torácico.Salt Lake City (2011)
Presentan una clínica muy diversa, pudiendo ser asintomáticos o producir fiebre, dolor, irritabilidad, pérdida de peso, hipertensión, estreñimiento y síntomas respiratorios (4). La extensión epidural o intradural puede provocar compresión de la médula espinal (5). Su pronóstico es heterogéneo, desde la regresión espontánea hasta un curso agresivo y fatal (6). El 35% de pacientes presentan diseminación ganglionar regional en el momento del diagnóstico y el 50% de pacientes debutan con enfermedad metastásica, principalmente en hueso, médula ósea e hígado (7).
La valoración inicial incluye la clínica de presentación, exploración física, pruebas de imagen, análisis de laboratorio, evaluación de la médula ósea y biopsia del tumor. Se han propuesto diferentes sistemas de estadificación tumoral. En 2009, el Grupo de Trabajo del Grupo Internacional de Riesgo del Neuroblastoma (INRG) propuso el Sistema de Estadificación del Grupo Internacional de Riesgo de Neuroblastoma (INRGSS) (7). Esta estadificación se emplea actualmente, y valora la extensión y resecabilidad tumoral en función de los Factores de Riesgo Definidos por Imagen (IDRF), establecidos mediante Resonancia Magnética (RM) o Tomografía Computarizada (TC) (3,8). La RM es la técnica de elección ya que permite valorar la invasión medular y muestra mayor contraste entre tejidos. La extensión local y la presencia de metástasis se evalúan mediante biopsia de médula ósea y estudios de Tomografía Computarizada por Emisión de Fotón Único (SPECT) con metaiodobenzilguanidina [123I]MIBG (9) (Figura 2). La biopsia del tumor permite llevar a cabo estudios biológicos y genéticos, siendo la ampliación del oncogén MYCN un factor asociado con una progresión acelerada de la enfermedad (10,11).
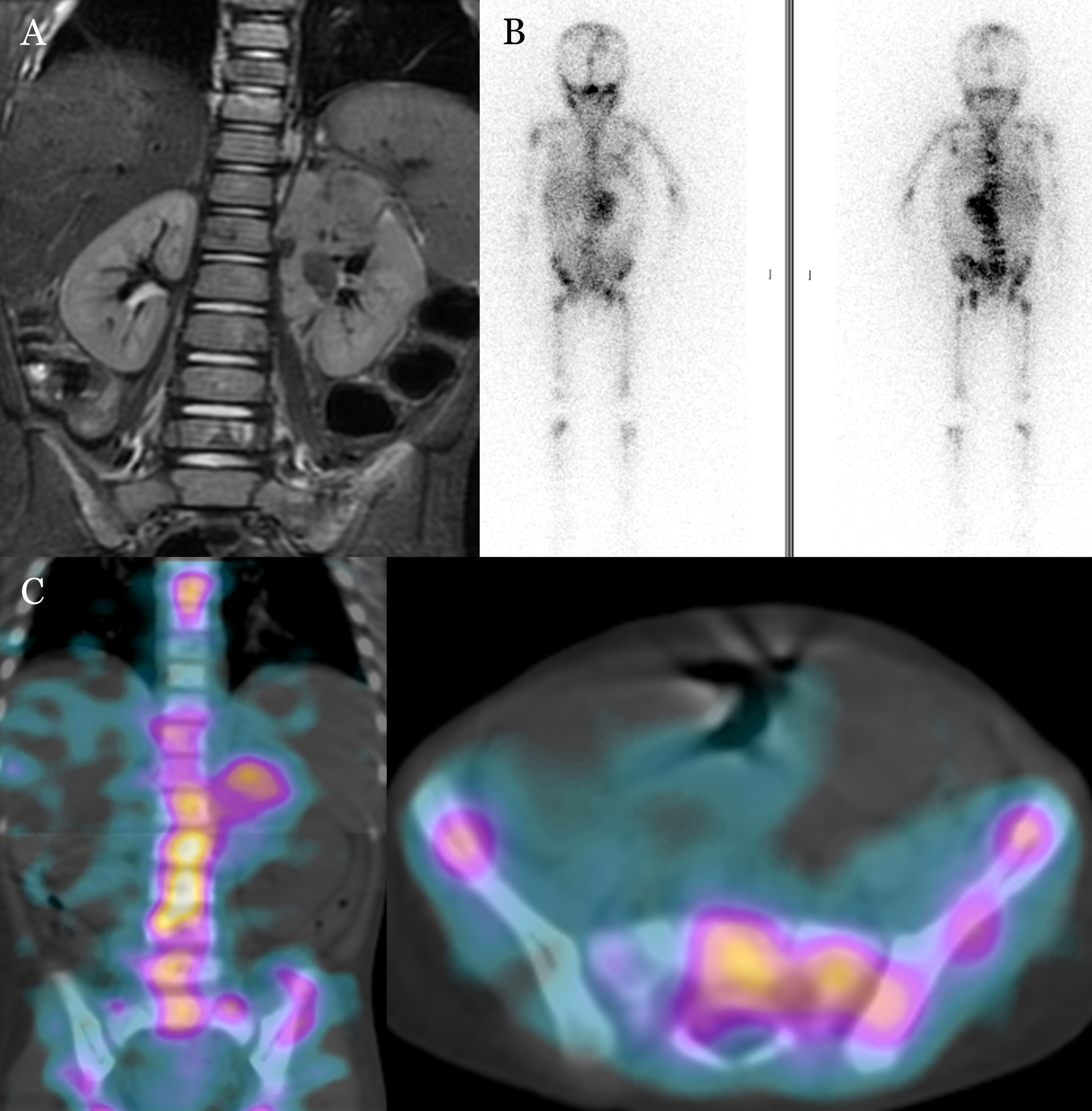
local y a distancia del tumor. (A) Resonancia Magnética en plano coronal que permite la evaluación de la extensión local de un tumor suprarrenal izquierdo. (B y C) Evaluación de la extensión a distancia mediante gammagrafía planar de cuerpo completo con [123I] MIBG (fila superior izquierda) y planos tomográficos SPECT (fila inferior) en orientación coronal (izquierda) y transversal (derecha).
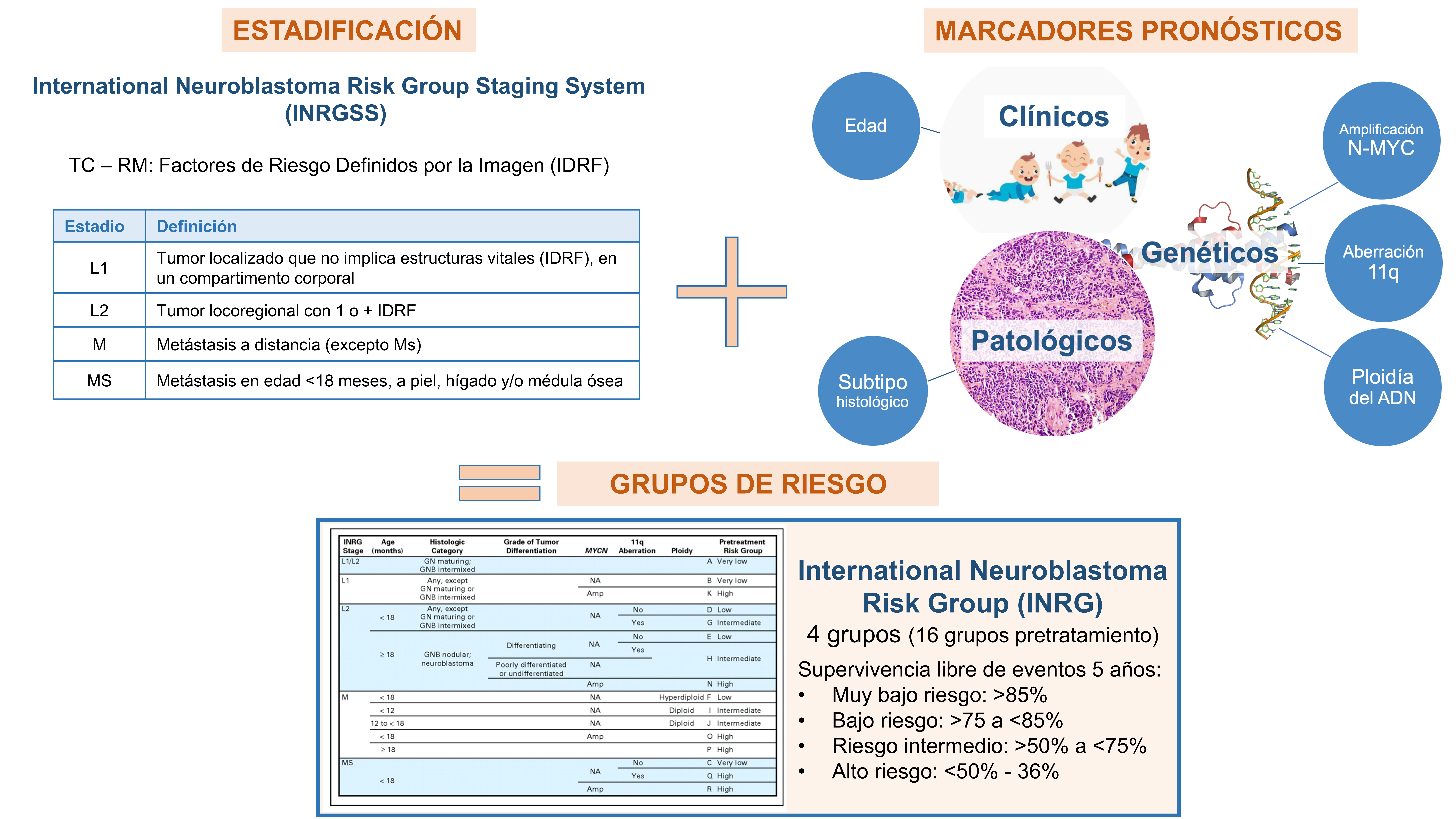
Los sistemas actuales de estadificación presentan limitaciones debido a la variabilidad en el comportamiento de los tumores, que en ocasiones resulta impredecible. No existe un consenso internacional sobre las variables pronósticas a evaluar para asignar grupos de riesgo y estratificar el tratamiento, y los factores seleccionados por varios grupos cooperativos expertos en neuroblastoma alrededor del mundo para definir el riesgo no son uniformes. Por ello, surge la necesidad de establecer nuevos biomarcadores adicionales para mejorar la precisión en la estratificación del riesgo tumoral.
Los biomarcadores de imagen son características estructurales y funcionales que proporcionan información fenotípica adicional a la lectura radiológica usual (12,13,14). La extracción de parámetros cuantitativos de las imágenes de RM podría constituir un biomarcador de imagen potencial en pacientes con neuroblastoma para mejorar la capacidad de estratificación del riesgo y la predicción de la agresividad tumoral, permitiendo una mejor tipificación de la enfermedad en cada paciente.
Varios estudios han demostrado que la radiómica puede contribuir en la caracterización de tumores (15,16). Sin embargo, la estabilidad y reproducibilidad de las características son limitaciones clave en la generalización de este tipo de estudios.
Para extraer los biomarcadores de imagen se necesita un proceso robusto y reproducible que incluya la eliminación de ruido, normalización de señal, remuestreo del vóxel y segmentación tumoral previos a la extracción radiómica y su integración con variables clínicas para obtener predicciones biológicas (14,17,18) (Figura 4). La segmentación manual del tumor presenta una amplia variabilidad intra e interobservador (19,20). La segmentación automática con aprendizaje profundo suele basarse en modelos de redes neuronales convolucionales como la red U-Net (21,22).
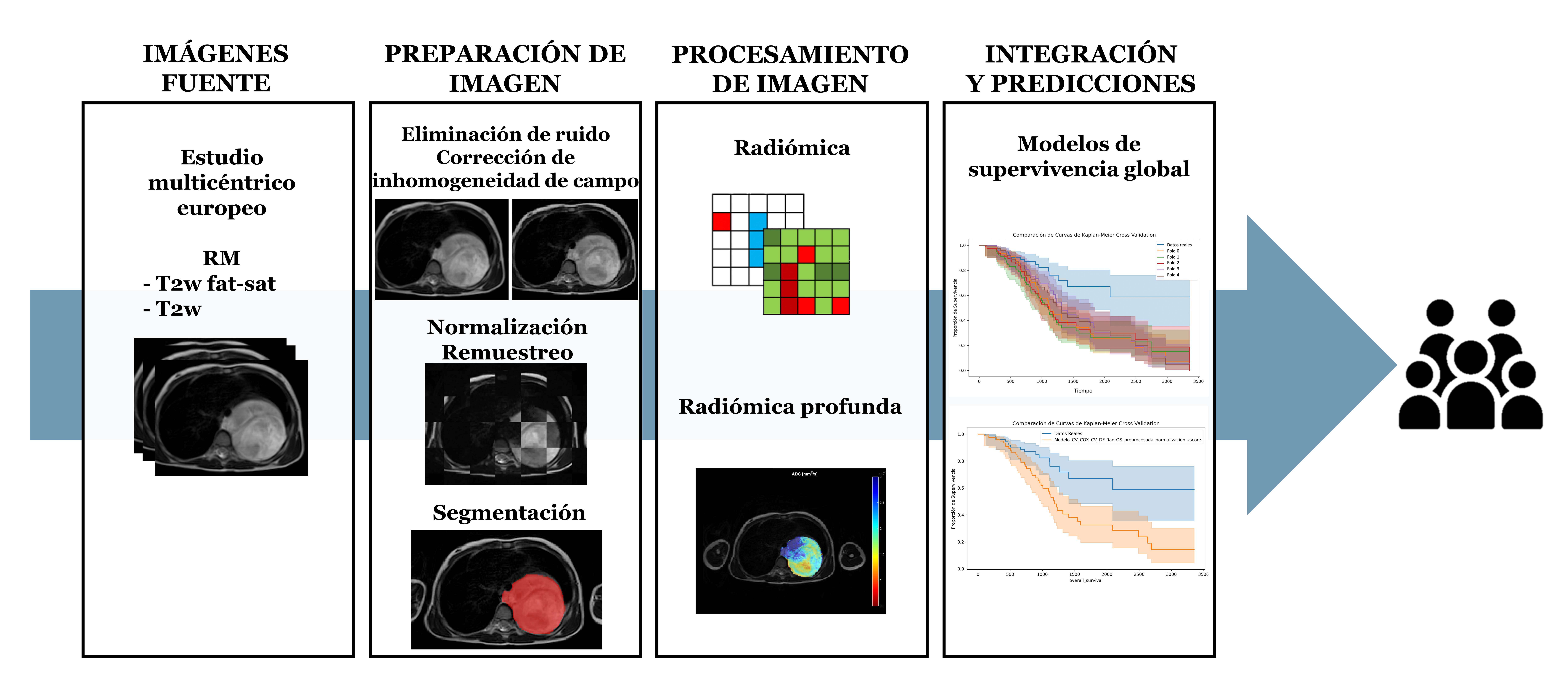
El objetivo del estudio es resumir nuestra experiencia en el análisis de las imágenes de RM en tumores neuroblásticos para determinar la agresividad tumoral considerada como la capacidad del tumor de extenderse en vecindad y a distancia, y de acortar la expectativa de vida.
MATERIAL Y MÉTODOS
Estudio de cohorte retrospectivo, observacional y analítico, multicéntrico, con la finalidad de crear modelos predictivos. Entre mayo de 2019 y junio de 2023 se reclutaron pacientes pediátricos con diagnóstico final de tumor neuroblástico para incluirlos en el proyecto europeo PRIMAGE (PRedictive In-silico Multiscale Analytics to support cancer personalized diaGnosis and prognosis, Empowered by imaging biomarkers) (23).
Se incluyeron 508 pacientes separados en un grupo de entrenamiento (392 pacientes) y otro de validación (116 pacientes) del modelo de supervivencia generado. Todos los pacientes disponían de una RM del tumor al diagnóstico. Todos los datos se seudonimizaron usando el European Patient Identity Management (EUPID) (24). Se recogieron variables clínicas, analíticas y de imagen atendiendo a las recomendaciones del INRG para la estratificación de riesgo de pacientes con neuroblastoma (7). Las pruebas de RM se almacenaron en la plataforma del proyecto PRIMAGE, una adaptación específica de QUIBIM Precision® (25). Todo este trabajo está basado en la memoria científica del proyecto PRIMAGE y en la Tesis Doctoral de uno de los autores (DVC).
Preparación de las imágenes
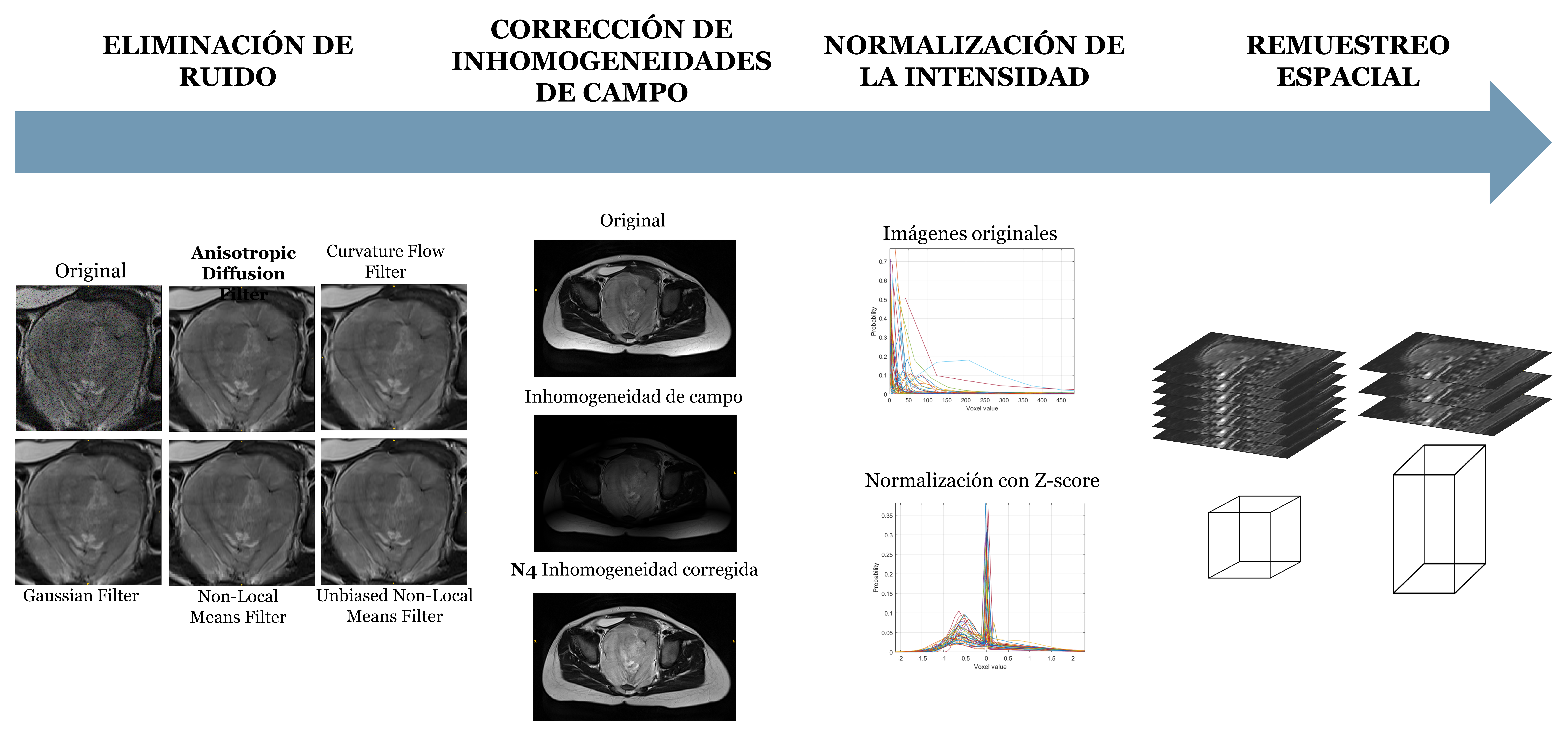
Se emplearon las imágenes potenciadas en T2 obtenidas con secuencias T2 spin echo (T2wSE) o T2* eco de gradiente (T2*wGE), con o sin supresión grasa (FS/STIR), en el plano transversal. Con la finalidad de reducir la variabilidad en los equipos y parámetros de adquisición se procesaron las imágenes mediante un Filtro de Difusión Anisotrópica (ADF) para reducir el ruido, corrección de la heterogeneidad en la distribución de la señal con filtro N4, normalización de señal mediante Z-score, y armonización espacial del tamaño de los vóxeles (Figura 5).
Segmentación y Radiómica
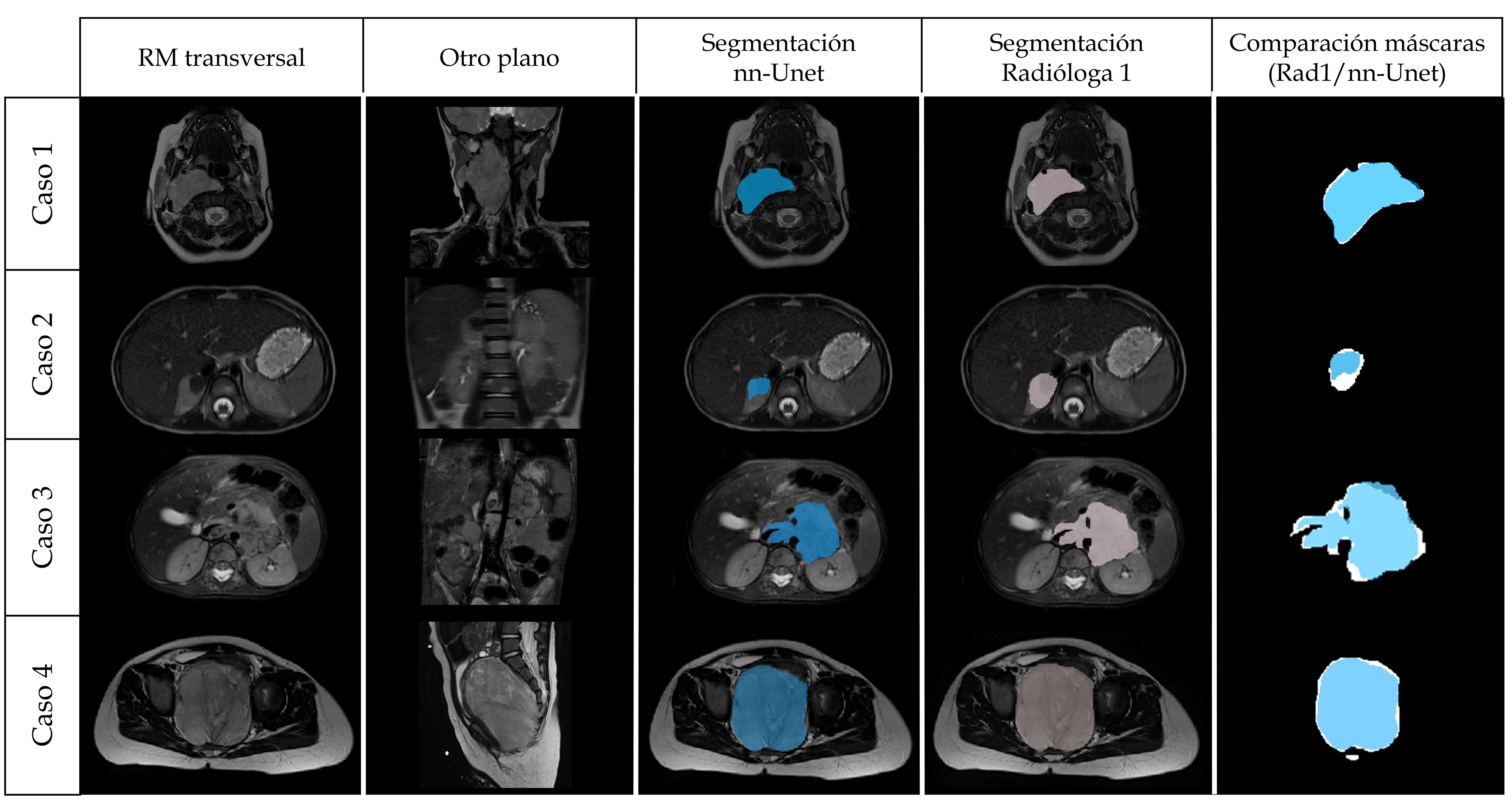
Empleando como patrón de referencia las máscaras de segmentación manual realizadas por radiólogos expertos con criterios restrictivos (incluyendo ganglios en contacto con el tumor, pero excluyendo vasos, órganos vecinos y lesiones a distancia), se entrenó una red neuronal convolucional 3D basada en el aprendizaje profundo denominada nnU-Net (26). La red fue entrenada con una estrategia de validación cruzada (27). La calidad de la segmentación automática se evaluó con el Coeficiente de Similitud de Dice (CSD) (28), como índice de superposición espacial y métrica de validación de la reproducibilidad (29). Se consideró también la proporción de falsos positivos (FP) y de falsos negativos (FN) (27).
Las características radiómicas se extrajeron utilizando la herramienta Pyradiomics (30). Para evaluar la reproducibilidad de la radiómica se utilizó el coeficiente de correlación de concordancia (CCC). El CCC se clasificó como excelente (si ≥ 0.90); bueno (0.75–0.89); moderado (0.50–0.75); o bajo (<0.50).
Modelos predictivos de supervivencia
Se empleó un modelo de Cox para la predicción de supervivencia. Para su explicabilidad se empleó la estrategia SHAP (SHapley Additive exPlanations), que permiten representar la contribución relativa individualizada de cada variable de entrada a la predicción de supervivencia de un paciente. Estos valores se calculan evaluando cómo cambia la predicción del modelo al considerar diferentes combinaciones de variables (31). En este estudio se obtuvieron las variables más importantes empleadas en el modelo de Cox a partir del conjunto de datos de validación. La evaluación de modelos de supervivencia se realizó con el índice de concordancia C-índex, que mide la proporción de pares de eventos y no eventos que se ordenan correctamente. Un valor de 0.5 indica que el modelo no tiene capacidad de discriminación mejor que al azar, mientras que un valor de 1.0 indica una discriminación perfecta. También se empleó el AUC-ROC para medir la capacidad del modelo para clasificar correctamente los eventos de supervivencia en los diferentes umbrales de probabilidad.
RESULTADOS
Se incluyeron 508 pacientes con tumores neuroblásticos y edades comprendidas entre el nacimiento y los 18 años (media de edad, 29±34 meses), con 243 niñas y 265 niños. La histología del tumor fue neuroblastoma (n=455), ganglioneuroma (n=19) y ganglioneuroblastoma (n=34), siendo la localización abdominopélvica (415 casos; de ellos 250 se situaban en glándula suprarrenal, 129 abdominales y 36 con localización pélvica) o cervicotorácica (93 casos; de los que 62 eran torácicos y 31 cervicales).
Segmentación
El estudio comparativo de la segmentación manual realizada por radiólogos en 132 secuencias de RM mostró una alta concordancia con un CSD de 0.969 (±0.032 IQR). Los modelos de segmentación automática obtuvieron una mediana de CSD de 0.965 (±0.018 IQR) y una mediana de AUC ROC de 0.981 (±0.010 IQR) (27). No hubo diferencias de calidad de segmentación según la ubicación, el campo magnético, la edad o el volumen tumoral (Figura 6). Los casos segmentados manualmente requirieron un tiempo medio de 56 minutos por caso, mientras que el tiempo medio necesario para obtener cada máscara con el modelo automático fue de 10 segundos. Todas las segmentaciones fueron validadas visualmente, y se realizaron ediciones y ajustes manuales de las máscaras automáticas cuando fue necesario (12 casos fueron editados, incluyendo 92 cortes). El tiempo medio para realizar estos procesos fue de 4.08 minutos (±2.35 DE).
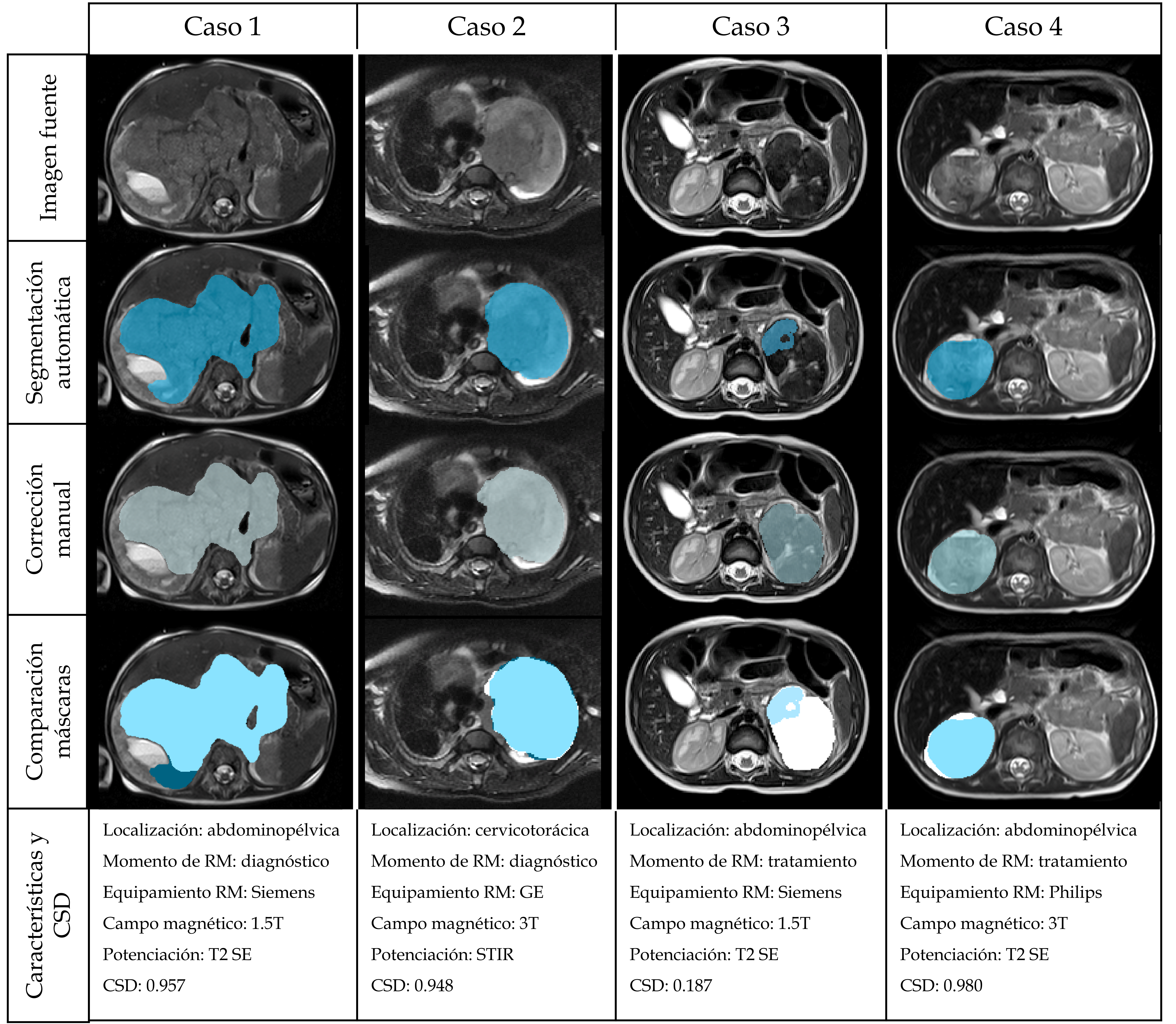
La validación independiente de la herramienta de segmentación automática sobre 535 secuencias diferentes de 300 pacientes con RM potenciadas en T2/T2* (Figura 7) tuvo un excelente acuerdo, con una mediana del CSD de 0.997 (±0.056 IQR) y el AUC ROC de 0.930. Solo el 14% (74 casos con un CSD < 0.8) requirió edición manual, y únicamente en el 6% (18 casos) la herramienta no detectó el tumor. No hubo diferencias por la ubicación, el momento del estudio, el campo magnético ni la ponderación de la secuencia, con resultados similares para todos los subgrupos (CSD de 0.995 a 0.999) excepto para los casos tras el tratamiento (CSD discretamente inferior de 0.902) (32).
Radiómica
Se extrajeron 107 características de radiómica de forma (n=14, describen la forma de la región de interés y sus propiedades geométricas), de primer orden o de histograma (n=18, describen la distribución de los valores de intensidad de los vóxeles sin tener en cuenta relaciones espaciales), y de segundo orden o textura (n=75, describen la relación espacial de brillos entre vóxeles vecinos). Las características de textura representan distintas matrices, incluyendo la coocurrencia de niveles de gris (GLCM, n=24), la dependencia de niveles de gris (GLDM, n=14), la longitud de segmentos consecutivos de píxeles con el mismo nivel de gris (GLRLM, n = 16), el tamaño de zona de niveles de gris (GLSZM, n=16), y la diferencia de tono de gris vecino (NGTDM, n=5).
Para evaluar la reproducibilidad y estabilidad de las características radiómicas tras el procesado de la imagen se generaron ocho series del conjunto de imágenes con diferentes modificaciones en su preparación y segmentación, evaluando el CCC con la serie basal (33). La eliminación de inhomogeneidades tuvo el menor impacto en la radiómica, con un 95% de características radiómicas estables al aplicar la normalización Z-score. La variabilidad de la segmentación (dilatación o erosión de las máscaras) mostró que el 74% de las características tenían un excelente acuerdo (CCC>0.90). Todas las variables de forma permanecieron estables y reproducibles en ambas series.
Predicción de Supervivencia
El modelo de supervivencia Cox, utilizando las 107 características radiómicas normalizadas mediante z-score, se aplicó a 508 pacientes, estratificados en conjuntos de entrenamiento (77%) y validación (23%). Se entrenó con validación cruzada con 5 subconjuntos para aumentar la robustez y generalización del modelo. La validación externa se llevó a cabo con otro conjunto de datos independientes con 116 pacientes. Se obtuvieron las curvas de supervivencia de Kaplan-Meier y se compararon los datos reales con las predicciones realizadas por el modelo.
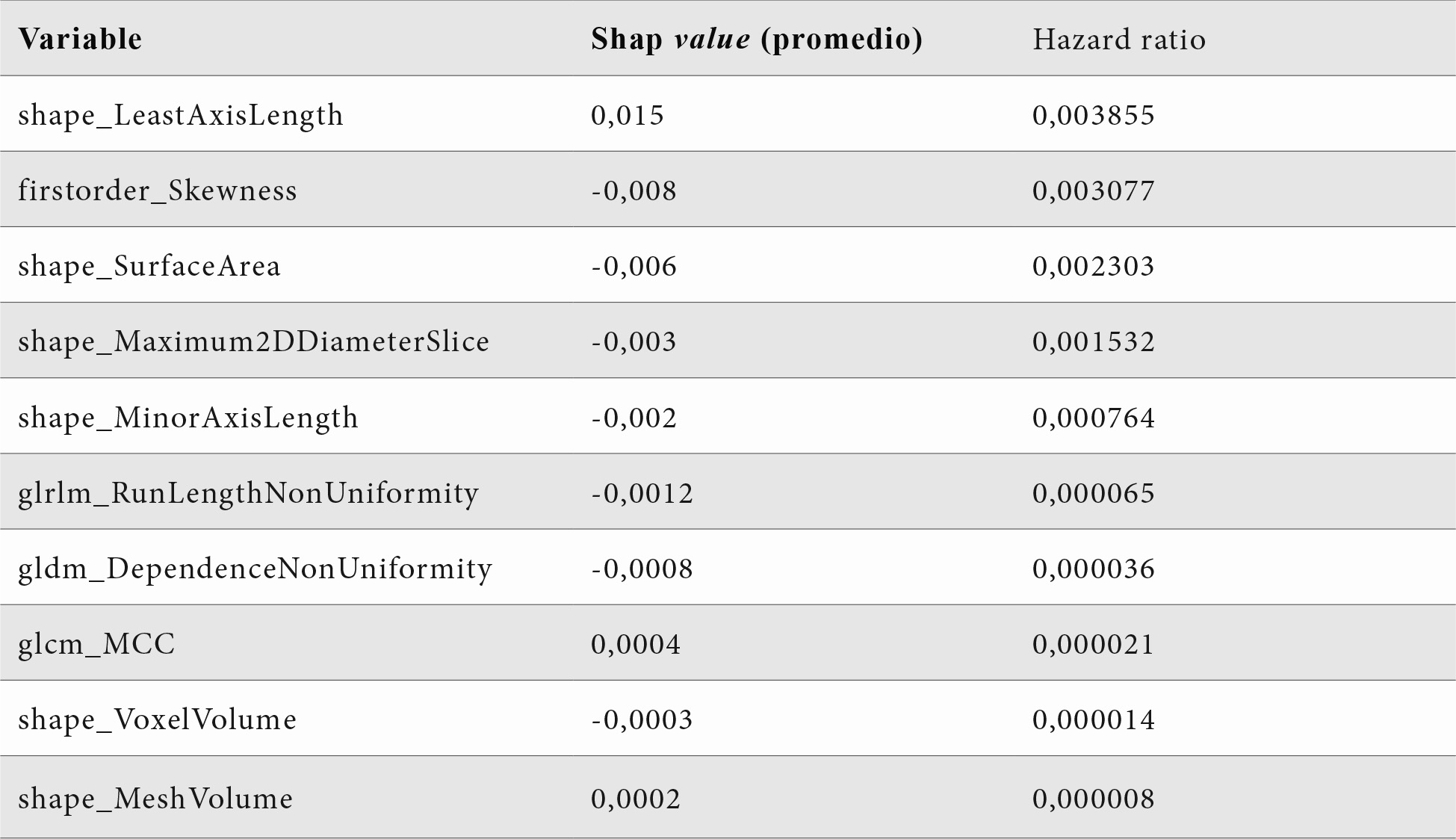
Se obtuvo la representación SHAP de las variables más influyentes (Figura 8). De las 20 variables más representativas, 7 fueron características de radiómica de forma, dos de primer orden y 11 de segundo orden. Se observó el impacto y la direccionalidad de cada variable de mayor representación. Por ejemplo, la variable de radiómica de forma Least axis length, que mide las variaciones en la longitud del eje más corto del tumor en 2D presenta un impacto positivo de la variable, lo cual implica que un mayor diámetro del eje más corto del tumor se asocia con un mayor riesgo de que suceda el evento (muerte). También, ante un valor mayor de la variable de radiómica de primer orden Skewness, referida a la asimetría de la distribución de las intensidades de los vóxeles pertenecientes a la región tumoral en torno al valor medio de la distribución (los tejidos altamente heterogéneos muestran una asimetría absoluta más alta que los homogéneos), existe mayor riesgo de fallecimiento del paciente. La variable de radiómica de segundo orden GLRLM (Run length non uniformity) mide la uniformidad en la distribución de las longitudes de las ejecuciones en la imagen. Un valor más alto de esta característica indica una mayor variabilidad en las longitudes de ejecución, lo que sugiere una textura más heterogénea en la imagen, y asocia a mayor riesgo de evento para el paciente.
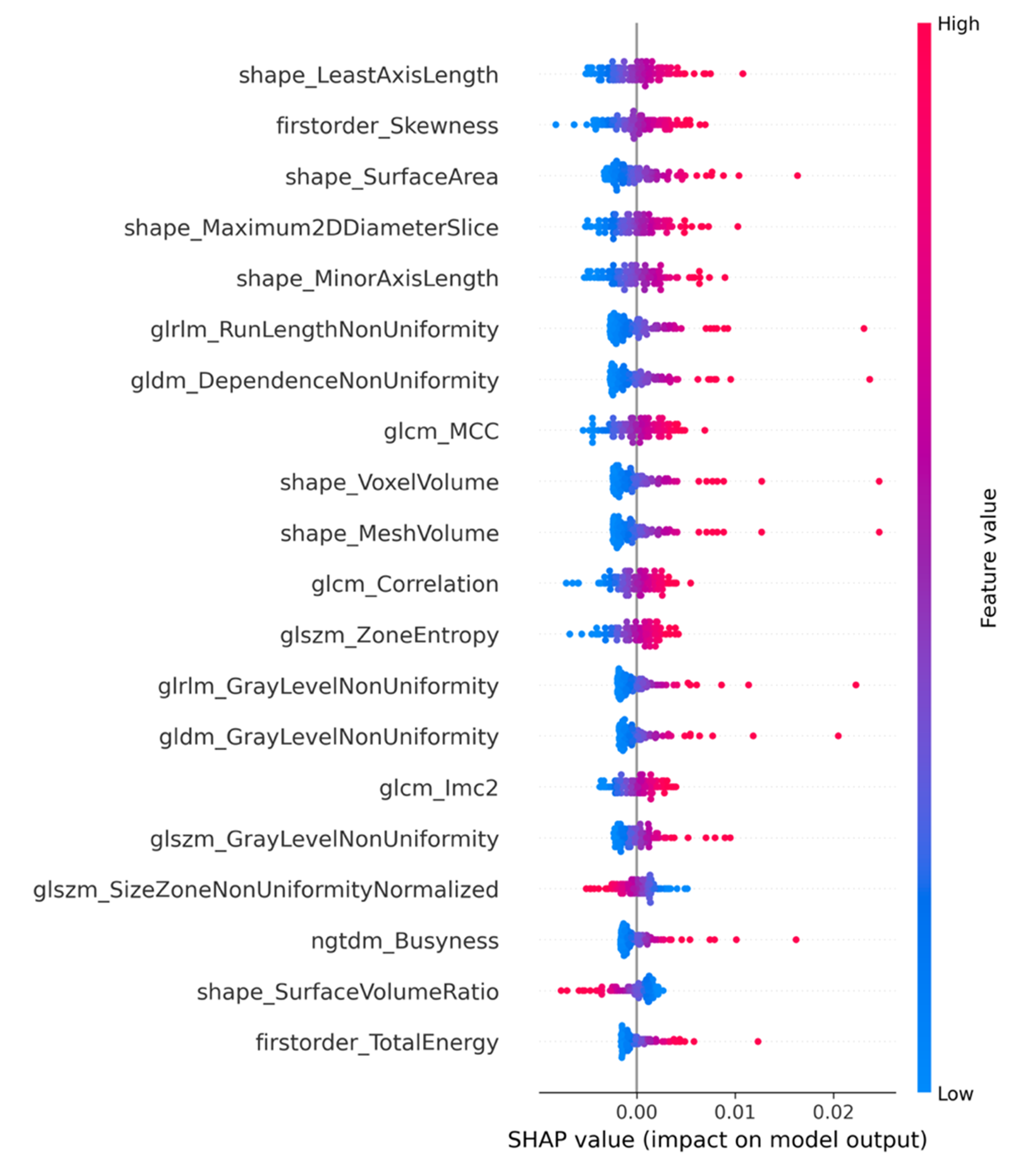
Las diez variables con un mayor valor predictivo en el modelo de supervivencia global fueron Shape least axis length, Skewness, Surface area, Maximum 2D diameter slice, Minor axis length, GLRLM Run length non uniformity, GLDM Dependence non uniformity, GLCM Maximum Correlation Coefficient, Shape Voxel Volume y Shape Mesh Volume. Estas características presentaron una relación positiva entre el valor de la variable y el riesgo de evento (Tabla 1). Las variables expuestas presentaron diferentes grados de reproducibilidad y estabilidad en los análisis realizados previamente. Estas diez variables de radiómica expuestas podrían considerarse como variables candidatas a biomarcadores de imagen para la predicción de supervivencia global en pacientes pediátricos con tumores neuroblásticos.
DISCUSIÓN
La imagen es fundamental en los tumores neuroblásticos para su detección, caracterización y predicción de la respuesta al tratamiento. Al tratarse de tumores muy heterogéneos, existe una necesidad clínica de mejorar la caracterización de su agresividad. Este trabajo resume la metodología computacional con biomarcadores de imagen, incluyendo los pasos de detección, segmentación tumoral y extracción de parámetros cuantitativos en RM, extrayendo características radiómicas que aportan una información complementaria a los sistemas de estadificación actuales. El proyecto PRIMAGE ha demostrado que los parámetros radiómicos mejoran los modelos de predicción de supervivencia, complementando los sistemas actuales. Los biomarcadores de imagen suponen una mejora diagnóstica y aportan un valor añadido en la predicción de la agresividad de los tumores neuroblásticos.
Se ha demostrado que la variabilidad al segmentar manualmente los tumores en las imágenes de RM es pequeña, con una alta concordancia entre observadores debido a las distintas habilidades individuales y mínimas sutilezas en los márgenes tumorales que representan un sesgo metodológico (34,35). Una solución de aprendizaje profundo nnU-Net detecta y segmenta los tumores neuroblásticos en las imágenes de RM con un rendimiento sólido (26,36). La arquitectura nnU-Net detecta y segmenta el tumor de forma automática, precisa y fiable, eliminando la variabilidad de la segmentación manual. Además, la solución minimiza los vóxeles falsos positivos que pertenecen a órganos o estructuras adyacentes, mejorando la precisión en el desarrollo de modelos radiómicos al no incluir estructuras normales (32). La segmentación automática minimiza el efecto de la variabilidad de la segmentación manual en la robustez de las características radiómicas extraídas (37), evitando el trabajo tedioso de la segmentación manual que se reduce a un ajuste final por el radiólogo, lo que reduce el tiempo requerido en un 93% y proporciona resultados reproducibles. Tan solo cabe mencionar que los cambios tumorales inducidos por la quimioterapia disminuyen discretamente la rentabilidad de la segmentación tumoral, aunque la diferencia no es estadísticamente significativa. La reducción del volumen tumoral con una peor delimitación de sus bordes después del tratamiento influye discretamente en el rendimiento de la segmentación automática, aunque el algoritmo automático es robusto y repetible incluso en este entorno.
El análisis de la reproducibilidad radiómica por el procesado para preparar la imagen o las variaciones discretas en las máscaras de segmentación demuestran que la eliminación de inhomogeneidades de señal tiene un impacto pequeño en las características radiómicas, aunque el remuestreo y la normalización alteran los resultados radiómicos presentando diferencias estadísticamente significativas. La normalización tiene un gran impacto en las características radiómicas de primer y segundo orden (33). Estos resultados confirman que la extracción de características radiómicas debe realizarse tras una normalización estandarizada de la señal de la imagen. La estabilidad de las características radiómicas de forma con pequeñas variaciones en la forma del tumor y la aplicación de diferentes filtros espaciales son pequeñas.
Las variables radiómicas han demostrado ser características relevantes para la predicción de la supervivencia global. Un estudio previo sobre radiómica en tumores neuroblásticos permitió diferenciar el neuroblastoma del ganglioneuroblastoma y ganglioneuroma en niños. Los resultados indicaron que las características de radiómica, combinadas con la edad del paciente en el diagnóstico inicial, proporcionan un método cuantitativo para distinguir estos tipos de tumores (38). Otros estudios han permitido el desarrollo de modelos de aprendizaje profundo basados en radiómica en imágenes de RM potenciadas en T2w para predecir la supervivencia global en pacientes oncológicos (39,40).
Las diez variables incorporadas al modelo de supervivencia presentaron una relación positiva en el modelo de supervivencia, de modo que una mayor longitud del eje más corto del tumor, una mayor asimetría en la distribución de los vóxeles, una mayor superficie tumoral, un aumento del valor máximo del diámetro tumoral en 2D, una mayor longitud del eje menor del tumor, una textura más heterogénea o un mayor volumen tumoral demostraron asociar un mayor riesgo de evento para el paciente, aumentando el riesgo de muerte al aumentar estos parámetros. Estas variables presentaron grados de reproducibilidad y estabilidad altos en los análisis realizados previamente. Por tanto, las diez variables de radiómica expuestas podrían considerarse como variables candidatas a biomarcadores de imagen para la predicción de supervivencia global en pacientes pediátricos con tumores neuroblásticos. La variable que mostró una mayor estabilidad en los análisis realizados previamente para evaluar la estabilidad y reproducibilidad de la radiómica fue GLRLM Run length non uniformity (no uniformidad de la longitud de ejecución). Esta heterogeneidad en la textura de la imagen se erige como un buen biomarcador no invasivo para la predicción de supervivencia global en pacientes con neuroblastoma.
Esta publicación tiene diferentes limitaciones que deben considerarse. El proyecto PRIMAGE se llevó a cabo en un conjunto de datos heterogéneo con parámetros de adquisición de las imágenes variables, equipos distintos, y en momentos temporales y ubicaciones diversas. Los modelos generados han solventado estas limitaciones inherentes a un estudio con datos del mundo real, lo que refleja la capacidad de sus resultados de transferirse a la práctica clínica para la tipificación fenotípica tumoral mediante radiómica. Además, el análisis solo incluyó imágenes potenciadas en T2, por lo que trabajos futuros podrían explorar el impacto de los análisis realizados en otras secuencias de RM, así como en otras pruebas de imagen como la TC o la SPECT.
Como conclusiones finales del proyecto PRIMAGE, el análisis de las imágenes de RM con algoritmos avanzados de procesamiento permite extraer características y descriptores de imagen cuantitativos con los que establecer predicciones sobre la agresividad biológica, entendida como supervivencia global, de los tumores de estirpe neuroblástica en pacientes de edad pediátrica. La precisión de la detección y segmentación automática de estos tumores neuroblásticos con la arquitectura de aprendizaje profundo nnU-Net supone una gran ventaja al reducir los tiempos para localizar y segmentar los tumores neuroblásticos con un gran porcentaje de éxito. Para garantizar la extracción reproducible de las características radiómicas debe estandarizarse el procesamiento de la imagen. La normalización de la imagen es el factor más influyente en la estabilidad de las características radiómicas. Las variables de radiómica son biomarcadores de imagen para la predicción de supervivencia en estos tumores.
AGRADECIMIENTOS
Agradecimientos: Parte de este trabajo está basado en la Tesis Doctoral de Diana Veiga-Canuto y en el Proyecto PRIMAGE (PRedictive In-silico Multiscale Analytics to support cancer personalized diaGnosis and prognosis, Empowered by imaging biomarkers), Grant Agreement 826494, Horizon 2020 SC1-DTH-2018-2020.
DECLARACIÓN DE TRANSPARENCIA
Los autores/as de este artículo declaran no tener ningún tipo de conflicto de intereses respecto a lo expuesto en el presente trabajo.
BIBLIOGRAFÍA
- ↑Bhatnagar SN. An audit of malignant solid tumors in infants and neonates. J Neonatal Surg. 2012; 1(1): 5.
- ↑Lonergan GJ, Schwab CM, Suarez ES, Carlson CL. From the archives of the AFIP: Neuroblastoma, ganglioneuroblastoma, and ganglioneuroma: Radiologic-pathologic correlation. RadioGraphics. 2002; 22(4): 911-934.
- ↑Brisse HJ, McCarville MB, Granata C et al. Guidelines for imaging and staging of neuroblastic tumors: Consensus Report from the International Neuroblastoma Risk Group Project. Radiology. 2011; 261(1): 243-257.
- ↑Irwin MS, Park JR. Neuroblastoma. Pediatr Clin North Am. 2015; 62(1): 225-256.
- ↑De Bernardi B, Pianca C, Pistamiglio P et al. Neuroblastoma with symptomatic spinal cord compression at diagnosis: Treatment and results with 76 cases. J Clin Oncol. 2001; 19(1): 183-190.
- ↑PDQ Pediatric Treatment Editorial Board. Neuroblastoma Treatment (PDQ®): Health Professional Version. 2024 Feb 13. In: PDQ Cancer Information Summaries [Internet]. Bethesda (MD): National Cancer Institute (US); 2002. Available from: https://www.ncbi.nlm.nih.gov/books/NBK65747/.
- ↑Cohn SL, Pearson ADJ, London WB et al. The International Neuroblastoma Risk Group (INRG) Classification System: an INRG Task Force Report. J Clin Oncol. 2009; 27(2): 289-297.
- ↑Monclair T, Mosseri V, Cecchetto G, De Bernardi B, Michon J, Holmes K. Influence of image‐defined risk factors on the outcome of patients with localised neuroblastoma: a report from the LNESG1 study of the European International Society of Paediatric Oncology Neuroblastoma Group. Pediatr Blood Cancer. 2015; 62(9): 1536-1542.
- ↑Matthay KK, Maris JM, Schleiermacher G et al. Neuroblastoma. Nat Rev Primer. 2016; 2: 16078.
- ↑Morgenstern DA, Pötschger U, Moreno L et al. Risk stratification of high-risk metastatic neuroblastoma: a report from the HR-NBL-1/SIOPEN study. Pediatr Blood Cancer. 2018; 65(11): e27363.
- ↑Ghosh A, Yekeler E, Teixeira SR, Dalal D, States L. Role of MRI radiomics for the prediction of MYCN amplification in neuroblastomas. Eur Radiol. 2023; 33(10): 6726-6735.
- ↑Martí-Bonmatí L, Alberich-Bayarri Á, Ladenstein R et al. PRIMAGE project: predictive in silico multiscale analytics to support childhood cancer personalised evaluation empowered by imaging biomarkers. Eur Radiol Exp. 2020; 4(1): 22.
- ↑Cozzi D, Bicci E, Cavigli E et al. Radiomics in pulmonary neuroendocrine tumours (NETs). Radiol Med (Torino). 2022; 127(6): 609-615.
- ↑Carbonell G, Kennedy P, Bane O et al. Precision of MRI radiomics features in the liver and hepatocellular carcinoma. Eur Radiol. 2022; 32(3): 2030-2040.
- ↑Tomaszewski MR, Gillies RJ. The biological meaning of radiomic features. radiology. 2021; 298(3): 505-516.
- ↑Lambin P, Leijenaar RTH, Deist TM et al. Radiomics: the bridge between medical imaging and personalized medicine. Nat Rev Clin Oncol. 2017; 4(12): 749-762.
- ↑Van Timmeren JE, Cester D, Tanadini-Lang S, Alkadhi H, Baessler B. Radiomics in medical imaging-“how-to” guide and critical reflection. Insights Imaging. 2020; 11(1): 91.
- ↑Sun M, Baiyasi A, Liu X et al. Robustness and reproducibility of radiomics in T2 weighted images from magnetic resonance image guided linear accelerator in a phantom study. Phys Med. 2022; 96: 130-139.
- ↑Joskowicz L, Cohen D, Caplan N, Sosna J. Inter-observer variability of manual contour delineation of structures in CT. Eur Radiol. 2019; 29(3): 1391-1399.
- ↑Bø HK, Solheim O, Jakola AS, Kvistad KA, Reinertsen I, Berntsen EM. Intra-rater variability in low-grade glioma segmentation. J Neurooncol. 2017;131(2): 393-402.
- ↑Feng F, Ashton‐Miller JA, DeLancey JOL, Luo J. Convolutional neural network‐based pelvic floor structure segmentation using magnetic resonance imaging in pelvic organ prolapse. Med Phys. 2020; 47(9): 4281-4293.
- ↑Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. En: Navab N, Hornegger J, Wells WM, Frangi AF, eds. Medical image computing and computer-assisted intervention – MICCAI 2015. Cham: Springer International Publishing; 2015. p. 234-241.
- ↑Veiga-Canuto D, Cerdá Alberich L, Fernández-Patón M et al. Imaging biomarkers and radiomics in pediatric oncology: a view from the PRIMAGE (PRedictive In silico Multiscale Analytics to support cancer personalized diaGnosis and prognosis, Empowered by imaging biomarkers) project. Pediatr Radiol. 2023; 54(4): 562-570.
- ↑Ebner H, Hayn D, Falgenhauer M et al. Piloting the European Unified Patient Identity Management (EUPID): Concept to facilitate secondary use of neuroblastoma data from clinical trials and biobanking. Stud Health Technol Inf. 2016; 223: 31-38.
- ↑QUIBIM Precision. https://cordis.europa.eu/project/id/778064.
- ↑Isensee F, Jaeger PF, Kohl SAA, Petersen J, Maier-Hein KH. nnU-Net: a self-configuring method for deep learning-based biomedical image segmentation. Nat Methods. 2021; 18(2): 203-211.
- ↑Veiga-Canuto D, Cerdà-Alberich L, Sangüesa Nebot C et al. Comparative multicentric evaluation of inter-observer variability in manual and automatic segmentation of neuroblastic tumors in magnetic resonance images. Cancers. 2022; 14(15): 3648.
- ↑Dice LR. Measures of the amount of ecologic association between species. Ecology. 1945; 26: 297-302.
- ↑Zou KH, Warfield SK, Bharatha A et al. Statistical validation of image segmentation quality based on a spatial overlap index1. Acad Radiol. 2004; 11(2): 178-189.
- ↑Van Griethuysen JJM, Fedorov A, Parmar C. Computational radiomics system to decode the radiographic phenotype. Cancer Res. 2017; 77(21): e104-107.
- ↑Lundberg SM, Lee SI. A Unified approach to interpreting model predictions. In: Guyon I, Luxburg UV, Bengio S et al. (Eds.) Advances in neural information processing systems 30. Red Hook, NY: Curran Associates; 2017. p.4765-4774.
- ↑Veiga-Canuto D, Cerdà-Alberich L, Jiménez-Pastor A et al. Independent validation of a deep learning nnu-Net tool for neuroblastoma detection and segmentation in MR images. Cancers. 2023; 15(5): 1622.
- ↑Veiga-Canuto D, Fernández-Patón M, Alberich LC et al. Reproducibility analysis of radiomic features from t2-weighted MRI after processing and segmentation alternations in neuroblastoma tumors. Radiol Artif Intell. 2024; 6(4): e230208.
- ↑Meyer CR, Johnson TD, McLennan G et al. Evaluation of lung MDCT nodule annotation across radiologists and methods. Acad Radiol. 2006; 13(10): 1254-1265.
- ↑Montagne S, Hamzaoui D, Allera A et al. Challenge of prostate MRI segmentation on T2-weighted images: Inter-observer variability and impact of prostate morphology. Insights Imaging. 2021; 12(1): 71.
- ↑Alves N, Schuurmans M, Litjens G, Bosma JS, Hermans J, Huisman H. Fully automatic deep learning framework for pancreatic ductal adenocarcinoma detection on computed tomography. Cancers. 2022; 14(2): 376.
- ↑Granzier RWY, Verbakel NMH, Ibrahim A et al. MRI-based radiomics in breast cancer: Feature robustness with respect to inter-observer segmentation variability. Sci Rep. 2020; 10(1): 14163.
- ↑Wang H, Chen X, Yu W et al. Whole-tumor radiomics analysis of T2-weighted imaging in differentiating neuroblastoma from ganglioneuroblastoma/ganglioneuroma in children: an exploratory study. Abdom Radiol. 2023; 48(4): 1372-1382.
- ↑Zhou Z. Artificial intelligence on MRI for molecular subtyping of diffuse gliomas: Feature comparison, visualization, and correlation between radiomics and deep learning. Eur Radiol. 2022; 32(2): 745-746.
- ↑Li G, Li L, Li Y et al. An MRI radiomics approach to predict survival and tumour-infiltrating macrophages in gliomas. Brain. 2022;145(3): 1151-1161.
ranm tv
Luis Martí-Bonmatí
Real Academia Nacional de Medicina de España
C/ Arrieta, 12 · 28013 Madrid
Tlf.: +34 91 547 03 18 | luis.marti@uv.es
Año 2024 · número 141 (03) · páginas 209 a 220
Enviado*: 18.06.24
Revisado: 02.07.24
Aceptado: 13.07.24
* Fecha de lectura en la RANM


